P(R) CALCULATION FROM PDB MODEL¶
P(r) 関数の観点からSAXSデータを検証することは、構造変化を理解または検出するための最良の方法である。 P(r) 関数がSAXS曲線の実空間表現であるにもかかわらず、 P(r) 関数は、最もノイズのない観測されたSAXS曲線の分解能が制限された視点である。しかしながら、関数自体は単純であり、特徴を解釈することは、既知の(ホモロジー)モデルからの P(r) 関数を重ね合わせることによって促進され得る。
ここで、 "P4P6_A4_2.mccd.dat" にPDBファイル single.pdb がロードされる。見てわかるように、データは広向 q (〜0.42Å -1 )にあたる。 PDBファイルをロードするには、ファイルを4つのパネルのいずれかにドラッグするだけである。 Scatter は自動的にファイルを P(r) 関数に基づくSAXSカーブに変換する。この計算には時間がかかることがあるため、待機すること。水分子はデフォルトで除外される。水分子を含めるには、 でフラグを変更できる(図2の赤い矢印を参照)。 SCÅTTER はPDBからの dmax を決定し、それを図1に示す。
図1
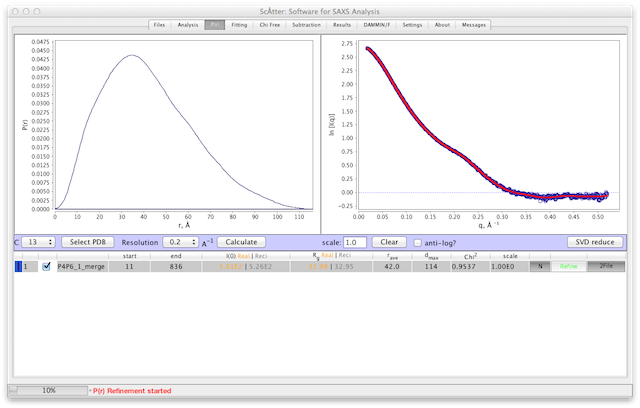
計算は、PDB内の全粒子に対して実行される。
図2
さらに、計算された P(r) 関数の分解能を設定することができる(デフォルト:0.4)。分子のサイズによっては、計算に時間がかかることがある。また SCÅTTER は、3D凸包を計算し、凸包の最も遠い2点を dmax として、入力分子の dmax を決定する。また、実験 P(r) を最適化して、データセットに適した115オングストロームの新しい dmax を決定した。
図3
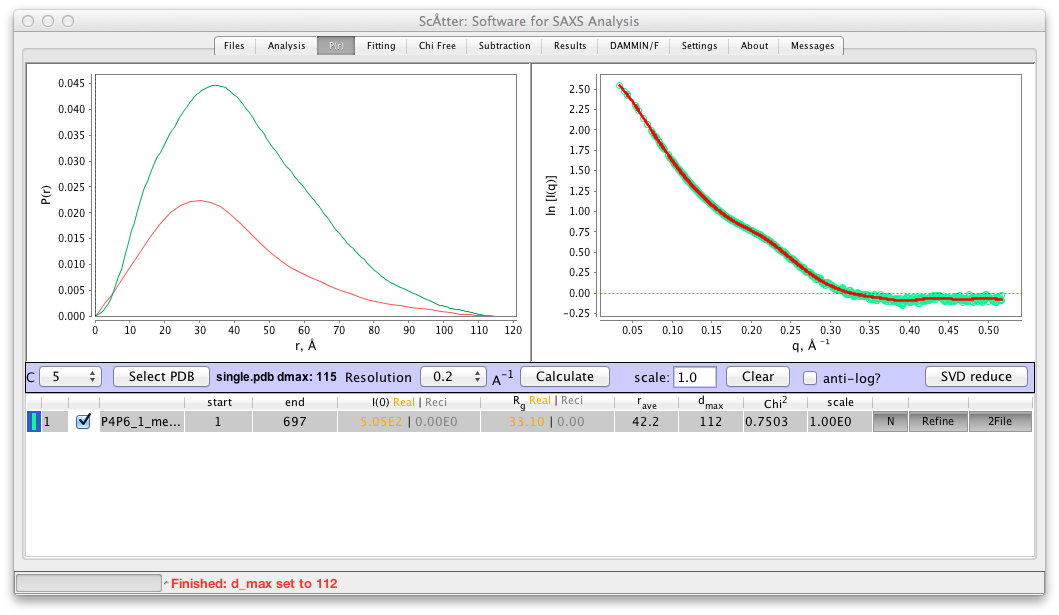
濃度、コントラスト、そして、実験強度のスケールなどの多くの理由からスケールはオフになる。したがって、本プログラムは設定可能なスケールを含めた、もし P(r) 関数が実験関数よりも小さい場合は、スケールを1.02に設定し、 キーを数回押して分布を高倍率にする。同様に、それが大きい場合は、スケールを0.98に設定し同様のことを行う。