ボックス2: MULCh の CONTRAST モジュールを用いたX線および中性子散乱コントラストの計算。¶
タイムチャート:¶
5分。
方法の概要。¶
MULCh (Modules for the analysis of small-angle neutron contrast variation data from bio-molecular assemblies)は、SASデータ解析を支援する一連のプログラムで構成されています。 MULCh の Contrast モジュールは、高分子系のX線と中性子の両方の散乱コントラスト(Δρ)を計算するために特化されています。 Contrast は、入力として散乱データを必要とせず、単にタンパク質、RNAまたはDNA配列と、溶媒小分子の濃度と原子式を組み合わせて使用する。この情報を使用して、Contrast は、高分子および溶媒のX線および中性子散乱長密度ρを計算し、これらの値を差し引いてサンプルのΔρを得る。
Contrast モジュールから得られたコントラスト値は、次の目的に使用できる。
高分子の分子量( MW )をSAXSおよびSANSの絶対スケール(cm-1 )に変換した \(I(0)\) から評価する
\[MW_{sample}=\dfrac{I(0)_{sample}N_A}{c_{sample}(\Delta\rho_{sample})^2}\]ここで、\(c_{sample}\) は濃度(\(g/cm^3\) )、\(v_{sample}\) は散乱粒子の偏比容 \(cm^3/g\) 、Δρはcm-2 単位のコントラスト、\(N_A\) はアバガドロ数である(
MW_from_absolute_scale.xlsx)。 タンパク質およびRNAについて、偏比容、\(\nu_{sample}\) は、NucProt の PSV および volume calculator 、http://geometry.molmovdb.org/nucprot/を使用して一次配列から計算することができ、または Contrast の出力から得ることができる。Contrast と NucProt の両方とも、その原子組成に基づいて高分子体積 V を計算することに注意のこと。- SAXSの場合
X線散乱コントラストを得ることにより小分子を溶媒に添加したときのΔρへの影響を評価する。散乱強度はΔρ2 に比例するので、高濃度の小分子の添加、 または電子密度の高い分子のサンプルへの添加は、溶媒と注目する高分子との間の電子密度の差、したがってΔρの差を減少させる。この情報は、放射線損傷を低減するため、サンプルに小分子(例えば、電子密度の高いポリオール、図2)が添加された場合のX線散乱強度(式2)への影響を評価するのに有用である。
- コントラストマッチングとコントラスト変調を伴うSANSの場合。
異なる%v/v \(\rm{{ }^2H_2O}\) 濃度で調製された溶媒中のサンプルの中性子散乱長密度およびコントラストを計算する。 これらの結果から、高分子と溶媒との間で交換される可能性のある酸性プロトンの割合を考慮に入れて、サンプル成分のコントラスト一致点を決定することができる(すなわち、 \(\Delta\rho=0\) になる溶媒中の%v/v \(\rm{{ }^2H_2O}\) ) (通常約90〜95%)。
注: Contrast モジュールで酸性 \(\rm{{ }^1H-{ }^2H}\) 交換値を変更して、サンプル成分のコントラスト一致点への影響を評価することができ、SANSのコントラストマッチング実験で使用する%v/v \(\rm{{ }^2H_2O}\) を決定するのに役立つます。 さらに、 Contrast からの V とΔρを使用して、SANSデータで成分がコントラスト一致する際の全体的な散乱強度大きさの変化を推定することができる。
- \(\rm{{ }^2H}\) ラベル付き部品を使用しているSANSの場合:ペプチド合成前。
もし交換不可能な高分子の重水素標識が考慮されている場合、 Contrast を使用して、異なるレベルの \(\rm{{ }^2H}\) 標識がサンプルの成分のコントラスト一致分離にどう影響するかを予測することができる。SANS実験を設定する前に Contrast を使用して、 目的のサンプル成分のマッチポイント分離を得るために、非交換可能な重水素化はどれぐらいのレベルが必要であるかを評価する。 これらの計算は、 \(\rm{{ }^2H}\) 標識された成分を調整する前に、生物学的に \(\rm{{ }^2H}\) 修飾されたペプチド合成のガイドとなる(ボックス3)。
- \(\rm{{ }^2H}\) 標識成分を使用するSANS:ペプチド合成後。
ペプチド質量フィンガープリンティング結果から実験的に決定された非交換可能な \(\rm{{ }^2H}\) のレベルを使用して、重水化で標識されたサンプル成分のSANSコントラストおよびマッチポイントを計算する(Box 3)。
- SANSの場合。
原子組成に基づいて、 \(\rm{{ }^1H_2O/{ }^2H_2O}\) 溶媒の質量密度を計算する。 デンシトメトリー測定値から実験的に決定された質量密度と比較して、溶媒の%v/v \(\rm{{ }^2H_2O}\) が正しいことを確認し、実験的なSANSコントラストを評価することができる。
オンラインツールは、必要な情報を入力するための基本的なコピー&ペーストまたは簡単なタイピング手順を使用します。 オフラインツールには、単純な入力テキストファイルが必要です(補足情報 Contrast_example_input.txt 、が例として提供されています)。 この単純なテキスト入力は、プログラムのオンライン版にアップロードすることもできます。 プログラムのオンラインバージョンとオフラインバージョンは、計算後に簡単なテキスト出力ファイルを生成し、オンラインまたはオフラインプログラムのいずれかによって再利用されます。 この手順全体は、図16を参照してください。
KinA:SDA 2:2 complex in 200mM NaCl, 50mM Tris, 150mM imidazole
7 # Number of contrast points
0.00 0.541 0.002 11.9 # D2O fraction, I(0), I(0) error, concentration (arb units.)
0.10 0.352 0.003 11.9
0.20 0.223 0.002 11.9
0.40 0.0745 0.002 26.9
0.80 0.19 0.001 11.9
0.90 0.332 0.002 11.9
1.00 0.537 0.001 11.9
3 # Number of things disolved in water
0.200 M NaCl 0.0 # Conc., M=molecule, formula, volume
0.050 M C4H11NO3 0.0 # " " Tris http://www.jtbaker.com/msds/englishhtml/t7111.htm
0.150 M C3H4N2 0.0 # " " Imidazole
0.95 # % of the exchangables that are accessible by the solvent
0.0 # % of the non-exch. protons in fragment 1 replaced by deuterons
1 # number of components, (next line) number of molecules, P=protein, sequence, volume
2 P GSHMTEELMLKSEKLSIAGQLAAGIAHEIRNPLTAIKGFLQLMKPTMEGNEHYFDIVFSELSRIELILSELLMLAKPQQNAVKEYLNLKKLIGEVSALLETQANLNGIFIRTSYEKDSIYINGDQNQLKQVFINLIKNAVESMPDGGTVDIIITEDEHSVHVTVKDEGEGIPEKVLNRIGEPFLTTKEKGTGLGLMVTFNIIENHQGVIHVDSHPEKGTAFKISFPKK 0.0
1.00 # % of the exchangables that are accessible by the solvent
0.85 # % of the non-exch. protons in fragment 2 replaced by deuterons
1
2 P GSMRKLSDELLIESYFKATEMNLNRDFIELIENEIKRRSLGHIISVSS 0.0
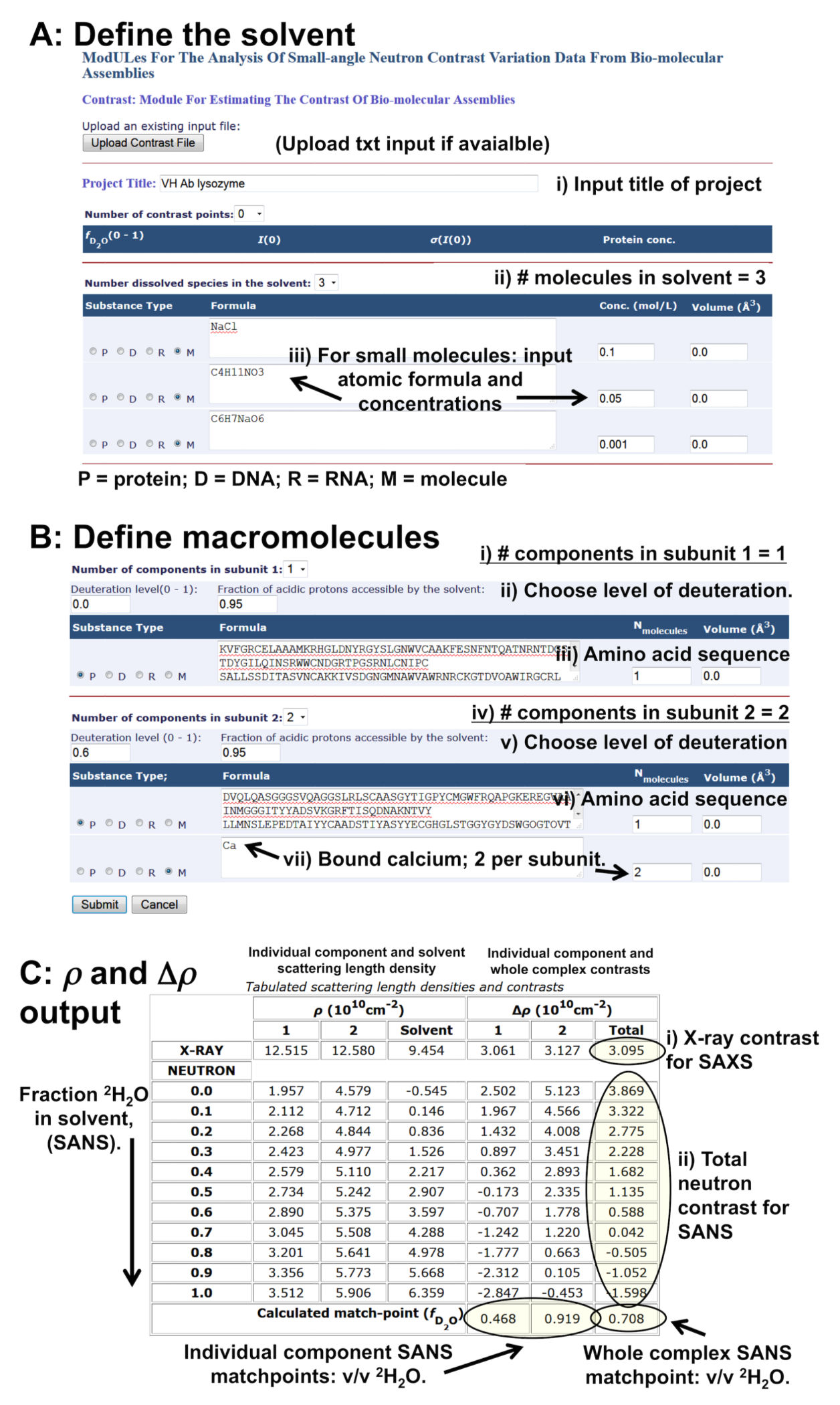
成分のX線および中性子のコントラストのMULChによる計算。 MULCh の Contrast モジュールのオンラインWebインターフェイスを示す。 A.溶媒の定義。 B.サンプルの巨大分子の定義。 この例では、2つのタンパク質の複合体があり、そのうちの1つは重水素化され、2つのカルシウムイオンを結合する。 C. SAXSとSANSのΔρを含むContrastの散乱長とコントラスト出力テーブル。 SANSの場合、Δρの大きさは、溶媒中の \(\rm{{ }^2H_2O}\) の割合を増加させるために計算される。 成分と複合体の完全一致点(Δρ= 0)を得るために必要な \(\rm{{ }^2H_2O}\) の割合が報告される。
装置¶
MULCh へのアクセス。¶
Contrast を含む MULCh パッケージ全体は、オフラインツール(説明書付き)としてダウンロードするか、オンラインでインタラクティブに使用できます:http://smb-research.smb.usyd.edu.au/NCVWeb/
材料¶
- 溶媒/緩衝液成分(原子式)の一覧とその濃度(mol/l)。
- 高分子の1文字のアミノ酸コードまたは1文字のDNA/RNAコード。
- 目的の高分子に結合した任意の小分子の原子式、例えば、金属イオン、補因子。
手順¶
- プロジェクトのタイトルを入力する。
- 溶媒を定義する。ドロップダウンメニューを使用して、溶媒中の溶解する化学種の数を選択します。これらは典型的には小分子、
Mである。各小分子について、Mが チェックされていることを確認し、各成分の原子式とその濃度(mol/L)を適切なボックスに入力する。 Contrast では水を含める必要はなく、自動的に 水溶液に対する小分子の添加による水の濃度の変化を計算する。 - 高分子を定義する(ステップ3-5)。ドロップダウンボックスを使用して、
Number of compenents in subunit 1を選択する。成分は、タンパク質、DNA、RNA または小分子であり得る。例えば、アミノ酸のみからなるタンパク質は、1つの成分を有する。金属タンパク質は2つの成分、すなわちタンパク質および結合金属を有する。 - サブユニットについて記載されている成分のタイプに応じて、
P =タンパク質、R = RNA、D = DNAまたはM =分子をチェックする。 高分子(P、R、D)については、配列全体の1文字コードを適切なボックスにコピーします。例えば、タンパク質については、Pをチェックし、 1文字のアミノ酸コードをボックスにコピーします。 DNAについては、Dをチェックし、順方向鎖および相補鎖の両方の1文字配列を確実にコピーし確認する。 必要に応じて、サブユニットにおける高分子の化学量論(N分子)を定義する。高分子(金属イオン、補因子など)に結合することが知られている 小分子の場合、Mを選択し、化学量論を含むことを念頭におきながら分子の原子式を入力する。例えば、1つのモノマーあたり2つのカルシウムイオンに 結合したタンパク質サブユニットは、`Number of components`= 2(すなわち、高分子およびカルシウム)、N分子、`P` (タンパク質)= 1およびN分子、M(カルシウム) = 2。 `number of components in subunit 2`を使用して、サンプルの2番目のサブユニットを定義します。
重要
<重要なステップ>
グルコースイソメラーゼなど均一な複合体サンプル(ホモオリゴマー)では、 Contrast では単一の subunit とみなされます。 このような状況で、 Contrast がΔρ計算を完了するためには、 Number of components in subunit 1 を定義するために使用したのと同じ情報を Number of components in subunit 2 のそれぞれのボックスに貼り付ける。 例えば、四量体タンパク質の場合、:i) Number of components in subunit 1 ; ii)チェック P ; iii)モノマーのアミノ酸配列; iv) Nmolecules = 4 を設定する(あるいはGuinier近似または \(p(r)\) vs r 、四量体のアミノ酸配列を入力し、 Nmolecules = 1 に設定する)。同じ情報を Number of components in subunit 2 セクションにコピーする。
注釈
要するに subunit は普通の「サブユニット」の意味ではなく化学種と考えるべきである。
- (オプション)重水素化された成分を使用するSANSについては、高分子に取り込まれた非交換性 \(\rm{{ }^2H}\) の平均レベルを
Deuteration levelボックスに入力してください。fraction of acidic protons accesible to the solventBox (デフォルトでは0.95)を使用して、高分子と溶媒との間のプロトン-重水素交換の推定値を入れる。 - Volume( \(\AA^3\) )Boxに数字があることを確認します(0.0であっても)。 コンポーネントの体積がわかっている場合は、その体積のボリュームを入力します。 体積が不明の場合は、値を0.0のままにして、原子体積を自動的に計算します。
submitを押します。
補足:コントラスト変調実験に関する解説図¶
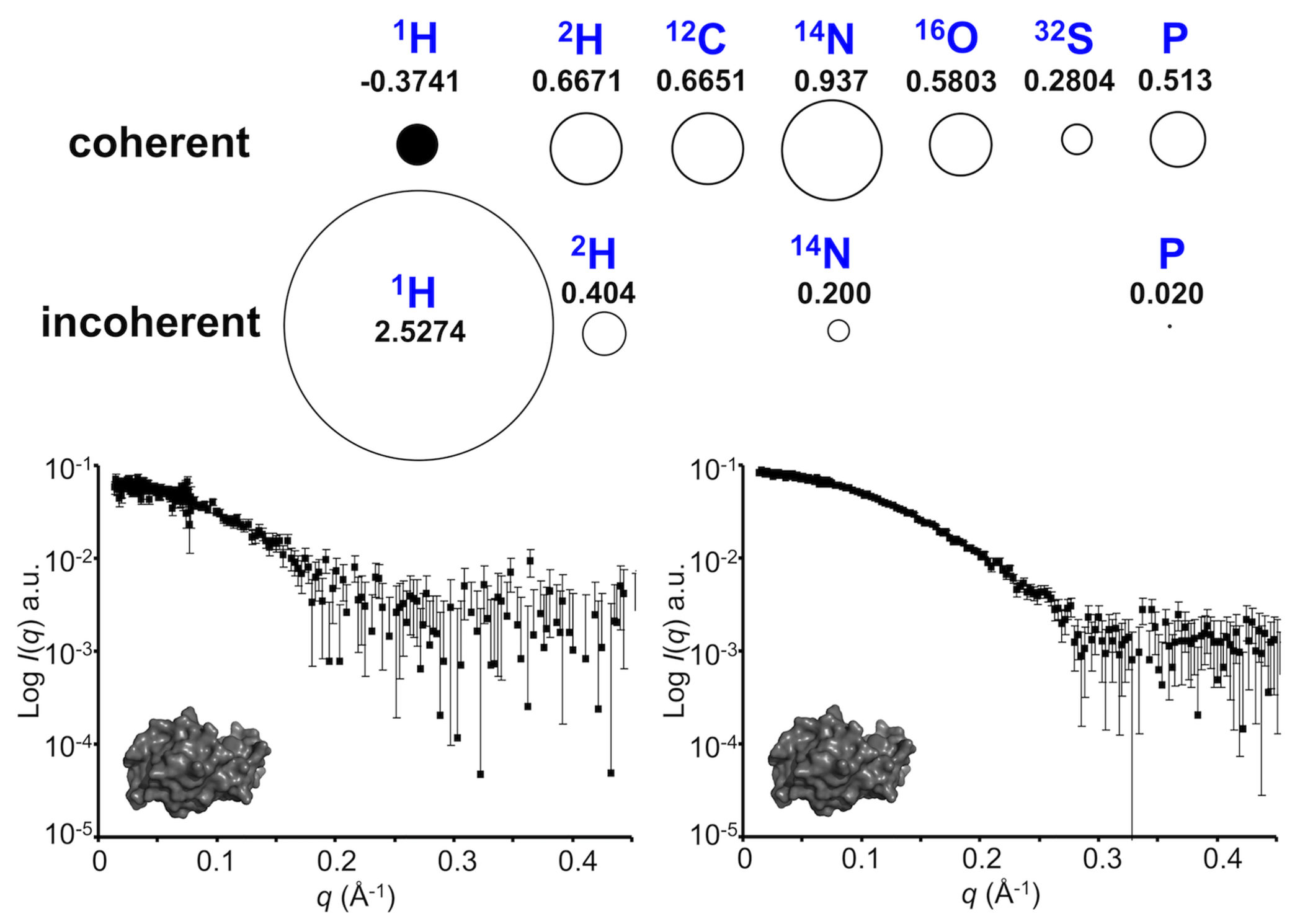
コヒーレントでインコヒーレントな中性子散乱。「生物」元素のコヒーレントおよびインコヒーレントな中性子散乱断面積( \(\sigma_c\) 、円として表示)およびそれぞれの中性子散乱長( \(b_c\,10^{-12}\rm{cm}\) ; \(\sigma_c=4\pi b_c^2\) )。 重水素および他の一般的に生じる生物学的同位体と比較して、 \(\rm{{ }^1H}\) は負のコヒーレント散乱長(黒丸で表される)を有する。散乱粒子体積内の相関距離から生じるコヒーレント散乱は、構造情報を抽出することができる散乱プロファイルを生成する。逆に、インコヒーレント中性子散乱は、形状/構造情報を抽出するために使用することはできず、SANSプロファイルのあらゆる角度にわたって「ノイズ」として寄与する。 \(\rm{{ }^1H}\) はかなりインコヒーレントな散乱長を有する。その効果は、100%v/v \(\rm{{ }^1H_2O}\) (左)におけるリゾチームからのSANS散乱によってが実証され、100%v/v \(\rm{{ }^2H_2O}\) (右)で収集された同じサンプルよりもかなりノイズを含む。 SANSデータは、同じ中性子波長、暴露時間、検出器距離、装置の形状、サンプル光路長、およびタンパク質濃度を用いてANSTO95のQuokka-SANS機器で収集した。中性子散乱長はSears、1992年による。
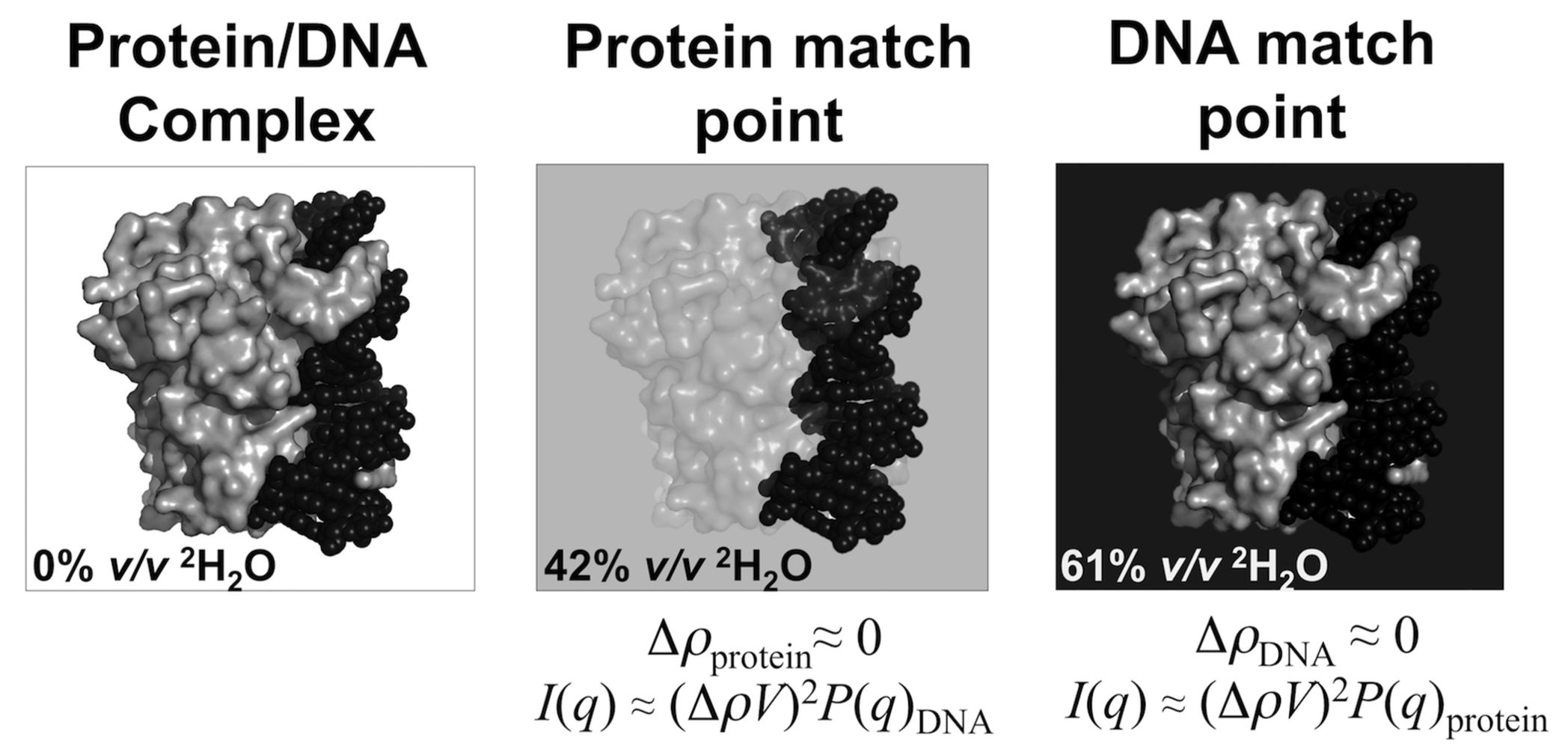
コントラストマッチングの原理 高分子複合体が異なる平均散乱長密度を有する個々の成分からなる場合、ある成分と同じ平均散乱長密度を有する溶媒中に複合体を配置することにより、その成分の散乱寄与をマッチアウトさせる(かき消す)ことが可能である。 ここにはタンパク質/DNA複合体(それぞれ灰色の表面および黒い球)が示されている。 例えば、中性子を用いて、複合体を約42% v/vの \(\rm{{ }^2H_2O}\) 、すなわちタンパク質のマッチポイントに入れると、測定されたSANSプロファイルのコヒーレント散乱はDNAによって支配され、そこから複合体を決定することができる。 %v/v \(\rm{{ }^2H_2O}\) を61%まで上昇させることは、SANSプロファイルがタンパク質からのコヒーレント散乱によって支配されるように、DNA散乱寄与と一致する。
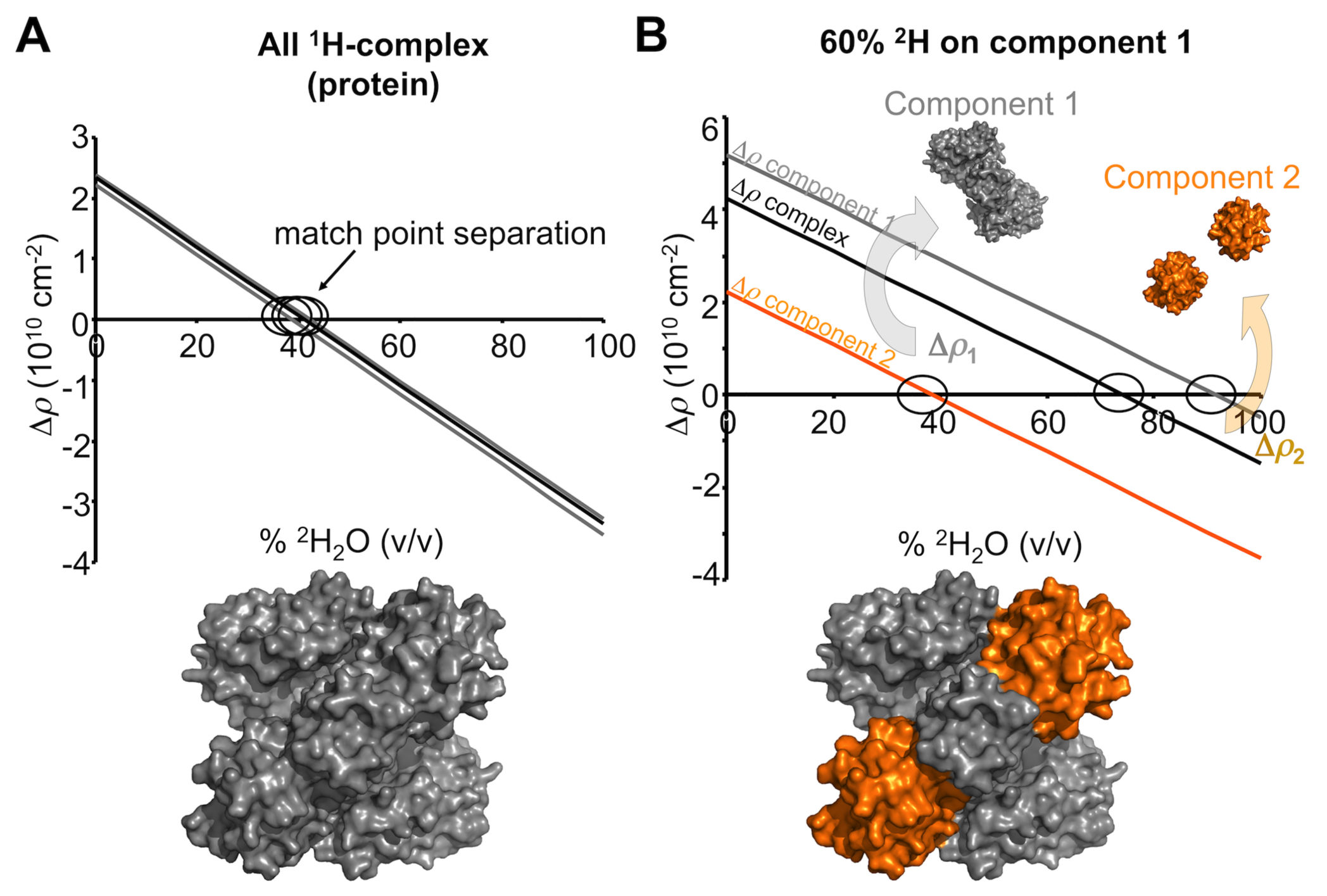
SANSのコントラスト変調実験におけるある成分の非交換重水素ラベル修飾の効果 A.タンパク質の単位体積あたりの平均 \(\rm{{ }^1H}\) にはほとんど差がないので、タンパク質-タンパク質複合体を含む成分について異なる%v/v \(\rm{{ }^2H_2O}\) で計算された中性子 コントラストは、ほとんど同一である(灰色および黒色の直線関係)。その結果、コントラスト変調実験によるSANSから復元された低分解能構造は、複合体全体の形状(グレーサーフェス表現)に反映される。 B.同位体標識非交換ジュウテリウムを有する成分は、コントラストと個々の構成要素のマッチングポイントと全体のマッチングポイントの分離に劇的な効果を与える。 この例では、成分1は、平均で60%交換不可能な \(\rm{{ }^2H}\) (灰色)であり、成分2は天然の \(\rm{{ }^1H}\) -タンパク質(オレンジ色)のままである。天然の \(\rm{{ }^1H}\) -タンパク質の散乱寄与が マッチングアウト(約40%v/v \(\rm{{ }^2H_2O}\) ;Δρ2=0;オレンジ色の線)したとき、散乱強度は成分1の \(\rm{{ }^2H}\) に由来し、その強度は、 \(\Delta\rho^2_1\) および \(V^2_1\) および \(P_1(q)\) に比例するであろう。%v/v \(\rm{{ }^2H_2O}\) を上昇させてさらに増加させた場合、ある点に到達する。 そのとき複合体全体のΔρがゼロ(約75% v/v;黒線)、散乱信号が例外的に弱くなる(本質的にインコヒーレント散乱および \(\rm{{ }^1H-{ }^2H}\) 交換からの散乱)。次第に、 \(\rm{{ }^2H}\) 成分1は高い%v/vの \(\rm{{ }^2H_2O}\) (約91%v/v \(\rm{{ }^2H_2O}\) ;Δρ1 = 0;灰色の線)でマッチングアウトし、 \(\rm{{ }^1H}\) 成分2からのコヒーレントな散乱寄与散乱寄与( \(\Delta\rho^2_2\) および \(V^2_2\) と \(P_2(q)\) )に比例する。コントラスト変調データのセットから、複合体全体、個々の成分の形状、複合体中の成分の向きを決定することが可能である。