タンパク質パラメータの計算¶
アミノ酸配列の取得¶
最初に、分子量を決定したいタンパク質のアミノ酸配列を用意しないといけない。 アミノ酸配列は基本的に1文字表記のテキストファイルである。以下では、配列が わからないとしてwebで検索して取得する。
NCBIのProetin database (https://www.ncbi.nlm.nih.gov/protein)にアクセスする。
検索したいタンパク質名を英語で入力する。その際、どの生物種由来かを明示すること。 この例では
bovine serum albuminと入力。この場合は、候補者リストの中からPDBファイルの配列を選択した。 PDBファイルは多くの場合、結晶化のためにHis-タグ等がついていたり、欠損している場合もあるので通常は回避する。 このPDBファイルは全配列が結晶化されているので問題ない。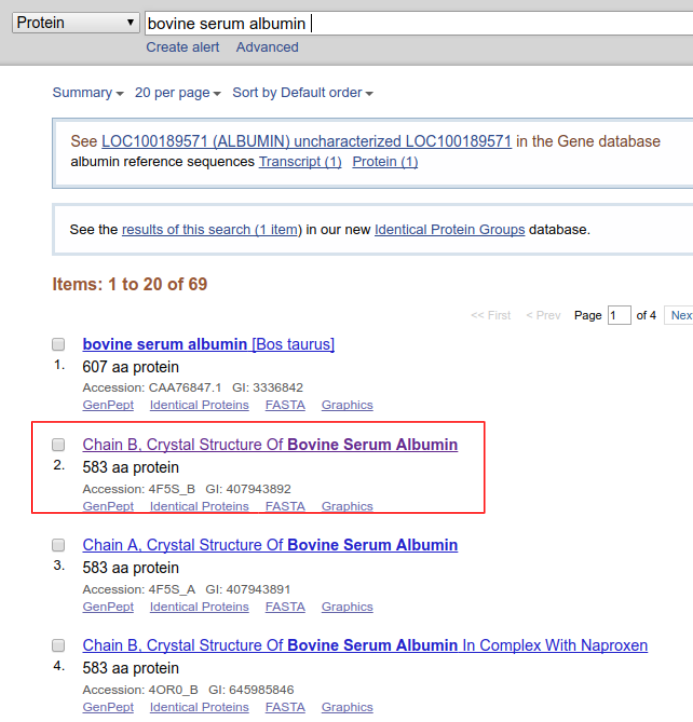
選択するとそのタンパク質に関わる様々な情報が表示される。
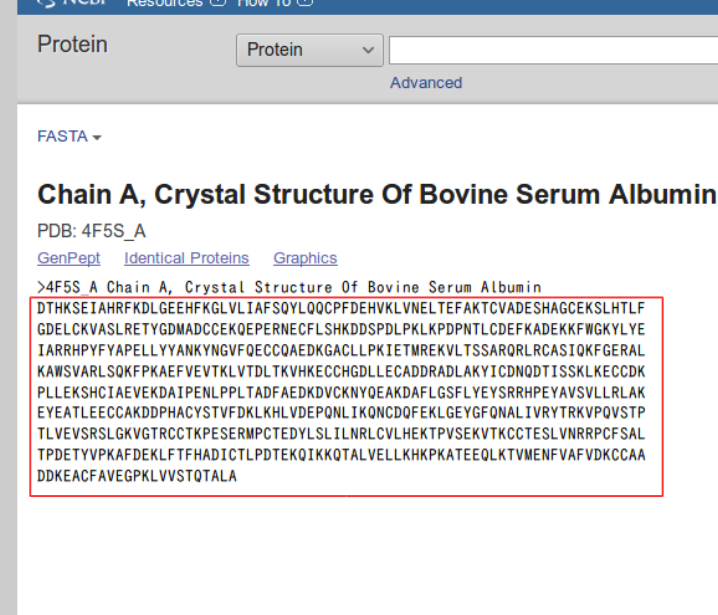
FASTAをクリックしてFASTAファイルを表示させる。 FASTAはアミノ酸配列の最も一般的なファイル形式である。
配列を別ファイルに保存する。 最初の行はファイルの説明でなので、配列部分のみをコピーする。
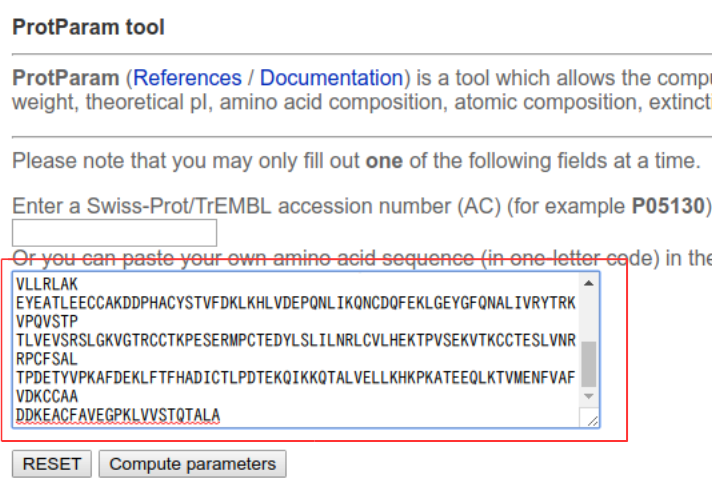
吸光係数の算出¶
吸光係数は PROTPARAM (http://web.expasy.org/protparam/)web serverを用いて計算する。
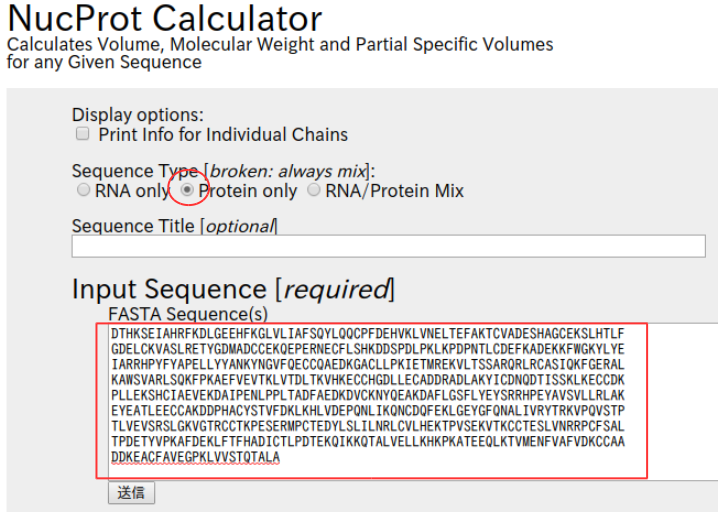
偏比容の算出¶
偏比容は NucProt (http://www.molmovdb.org/cgi-bin/psv.cgi)を使用して計算する。
BSAのパラメーター¶ Length 583 residues Volume 81383.4 A^3 Weight 66446.7 Da PSV 0.7376 mL/g \(\rm{A_{280nm}^{1mg/ml}}\) 0.646 pI 5.6