フィラメントタンパク質複合体の解析¶
次に、フィラメント解析の現状の手順について記載した。
手順としては、先程のチュートリアルで示したものと同様である。しかし、入力するパラメータ等は異なる。
重要
負染色データを扱うための設定
常に、
invert: | : uncheck |
|---|---|
xraypixel: | : unchek |
ac: | : 80 |
CTFの補正では、
lores: | : check |
|---|
にチェックを入れる。
手順の概要を以下に示す。
![// graphviz での作成の流れ
digraph G1 {
graph [size="6,6"];
node [shape=box];
a [label="EMAN2の起動"];
// b [label="データの取り込み"];
c [label="電顕画像から粒子の抽出"];
c1 [label="スタック画像の取り込み"];
d [label="データのマニュアル補正"];
e [label="データセットの編成"];
e2 [label="",shape=circle];
a-> c [label="orig_micrographs/*.dm3"];
// b-> c [label="micrographs/*.hdf"];
c -> c1 [label="*_helix_ptcl.hdf"];
c1-> d [label="particles/*_helix_ptcl.hdf"];
d -> e [label="particles/*_helix_ptcl_ctf_flip.hdf"];
e -> e2 [dir=none];
{rank=same; a; e1;}
subgraph cluster1 {
label = "Modeling";
labelloc = "t";
labeljust = "l";
e1 [label="",shape=circle];
f [label="2Dクラス分類",shape=doubleoctagon];
g [label="クラス平均の取捨選択"];
h [label="初期モデル作成"];
f -> g [label="r2db#/classes*.hdf"];
g -> h [label="r2db#/good_classes.hdf"];
e1 -> f [label="sets/all_ctf_flip.lst"];
}
}](../_images/graphviz-bfefe4854eeb37ee862baa3024591e9574e9dbbf.png)
図 41 EMAN2 でのヘリックス解析の概要¶
データの準備¶
電顕画像の保存¶
まず、電顕で撮影を行った結果は、 dm3形式 で保存しておく。
注釈
保存するときのファイル形式について
tiff形式 で保存しておく場合だと、ヘッダー情報が欠落してしまうので、 DM3 、GATAN format で保存したほうが良さそう。
画像を確認する際には、 tif形式 のほうが扱いやすい。解析の際には、 dm3 が適している。なので、両方保存しておく。
保存方法は、 save as と save display as で保存。詳細は、電顕マニュアルに記載した。
電顕写真の情報は、 GATAN micrographs で、 より確認できる。
電顕画像のフィルタリング¶
EMAN2 のチュートリアルでは、クライオデータを解析することを想定しているので、 電顕画像の処理に若干の違いが出る。
負染色像の観察する際には、原則ジャストフォーカスではなくアンダーフォーカス側で観
察するものである。また、 CTF 関数で最初の 0 点が 2nm になるのは defocus 値が 100kv で
1100nm 、 200kv で 1600nm で負染色方ではこれより小さい値で測定を行う。
我々は、アンダーフォーカス側に 500~1000nm 程度ずらした条件で観察を行った。
defocus 値が 500~1000nm の場合は、 2nm 以内なのでシグナルの反転は起こらないため
CTF補正は行わない 。とされている。
しかし負染色データを単粒子解析する際には、電顕画像にCTF補正を行う必要があるみたい。 フィルタリング等に関しては、別に記載されているが、詳細な方法はよくわからない。 e2filtertool.py https://blake.bcm.edu/emanwiki/EMAN2/Programs/e2filtertool
EMAN2 の起動¶
プロジェクトマネージャーの起動¶
まず、 EMAN2 で電顕画像を処理するためにプロジエクトマネージャを起動する。
以下に示すように、解析する画像ファイルの入ったディレクトリで、 e2projectmanager.py と入力して、実行する。
$ e2projectmanager.py
すると、プロジエクトマネージャが起動する。
パラメータの設定¶
起動したら、はじめにプロジエクトのパラメータを設定する必要がある。
をクリック。
実際に解析をする際には、
電顕のパラメータを入力。
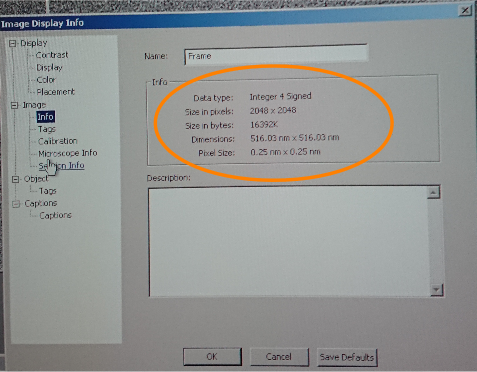
電顕のパラメータとしては、球面収差補正の値 CS 、1ピクセルの値 Apixs 等が必要
パラメータとしては、
質量: | : 1500kDa |
|---|---|
CS: | : 2.0 |
apixs: | : 2.53 |
Voltage: | : 120 |
ヒント
パラメータ値について
cs: | (球面収差補正値)は、電顕固有のパラメータとなっているので常に 2.0 。 |
|---|---|
apix: | 40k で撮影した場合には、 apixs=2.53 となっている。 |
Voltage: | 120kV で撮影したので 120 |
質量: | は画像パラメータではないようだが不明。仮に、一巻が400kDa程度として三巻がbox指定されるとして設定。 |
注釈
e2projectmanager.py のフローから外れた時
現状では、 e2helixboxer.py を使用すると e2projectmanager.py の流れから外れてしまう。 それ故、電顕画像一枚から粒子像を抽出した結果から初期モデルの構築を行っている。 画像の質、粒子の抽出の仕方にも依存してそう。
繊維像の抽出、スタック画像の作成 e2helixboxer.py¶
粒子の抽出、スタック画像の作成に関しては、プロジェクトマネージャーから e2boxer.py 実行するのではなく、 別でプログラムを呼び出して実行する。
- プログラムの起動
はじめに、繊維を抽出するプログラムである e2helixboxer.py を起動する。 起動は、電子顕微鏡写真の存在するディレクトリに新たにターミナルを開き、以下のコマンドを実行する。
$ e2helixboxer.py --gui orig_micrographs/*.dm3
また、以下のように実行することで、ディレクトリ内の電子顕微鏡画像を選択することも可能。
$ e2helixboxer.py --gui *.dm3
- 繊維の選択
電子顕微鏡画像より繊維を選択する。
繊維を長方形で囲むことで選択される。選択すると選択したヘリックスは Current Helix に表示され、
e2helixboxer の表に Boxed Helices の数が増える。
その際の操作方法を以下に示す。
| boxの作成 | 左クリックしてドラック |
| boxの移動 | boxの中央付近を左クリックしてドラック |
| boxの長さの変更 | boxの端を左クリックしてドラック |
| boxの削除 | Shiftを押しながら、左クリック |
ヒント
e2helixboxer.py の使用方法
また、繊維を選択する際の長方形のサイズを指定することができる。プロトフィラメントの選択に使用する箱の幅( Box width )の検討する必要があるかも。
画像では Box Width は 100 。
- スタック画像の作成
boxを指定したら、各画像ファイル毎にスタック画像として保存を行う。 e2helixboxer のコントロール画面で
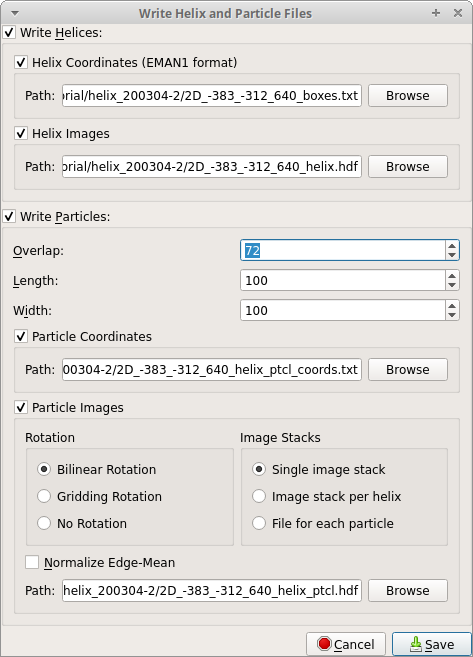
保存する際は、抽出したプロトフィラメントの周期パラメータ ( Overlap 、 Length 、 Width )を設定して保存を行う。
各パラメータは以下の定義となる。
保存することで、
- 繊維像の座標 :
(元画像ファイル名)_helix_ptcl_coords.txt - 各繊維像の画像 :
(元画像ファイル名)_helix_helix_#.hdf - スタック画像 :
(元画像ファイル名)_helix_ptcl.hdf
等がプロジェクトのルートディレクトリ保存される。
ヒント
e2helixboxer.py においてどのような「粒子」の単位で画像処理をしているのか
スタック画像ではよく分からないのでEMDBのXY投影像で比較すると particle length はほぼ3巻き(3ピッチ)で、 overlap は、2巻きであった。
e2helixboxer.py は、プロトマーではなく1巻きを粒子像として認識しているようであった
- スタック画像の取り込み e2import.py
次に、本来のデータ解析の流れに戻すために、 再度、プロジェクトマネージャーを起動して helixboxer.py で作成した スタック画像を取り込む作業を行う。
で
上で作成した複数の (元画像ファイル名)_helix_ptcl.hdf を取り込む。
particles というフォルダができ、その下に (元画像ファイル名)_helix_ptcl.hdf
がコピーされる。
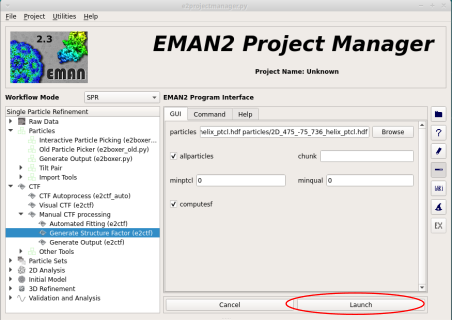
データのマニュアルCTF補正 e2ctf.py¶
- CTF補正を行う
で particles の下の (元画像ファイル名)_helix_ptcl.hdf を選択する。
オプションは、
| ac: | 80 |
|---|
にしておくこと。
この後、 で微調したほうがいいかもしれない
- 構造因子の出力
で、ファイルは上が選択されているのでこれはただ Launch をクリックすれば良い。
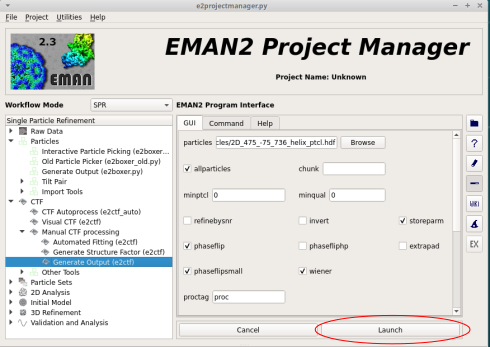
- CTF補正されたファイル出力
で、ファイルは上が選択されているのでこれはただ Launch をクリックすれば良い。
この操作により particle フォルダに (元画像ファイル名)_helix_ptcl_ctf_flip.hdf
などが生成される。
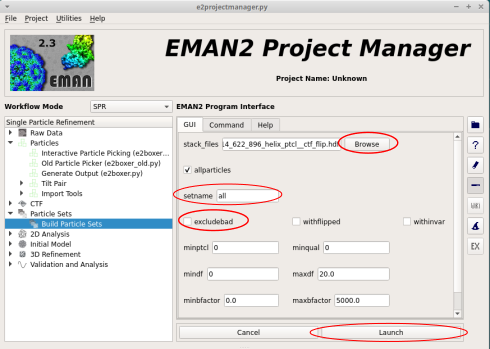
データセットの編成 e2buildsets.py¶
を選択する。
stack_files で
particlesのディレクトリにあるスタック画像(元画像ファイル名)_helix_ptcl_ctf_flip.hdfを選択する。
- 最初に出てくるファイルブラウザだと
*_ctf_flip.hdfが見えないのでファイルフィルタを外すと(元画像ファイル名)_helix_ptcl_ctf_flip.hdfが見える。
allparticlesをチェックし、excludebadチェックボックスを外す。
- 顕微鏡写真を手動で評価するときに低い品質値を指定しといた場合、
minqualボックスに5を入力することもできます。これにより、品質が5未満に設定された画像が除外できるセット名ボックスに任意の名前を入力する。ここでは、「
all」と入力Launch を押す。
これにより sets というフォルダでき、その下に all.lst 、 all__ctf_flip.lst 、 all__ctf_flip_small.lst
などが生成される。 __ctf というリストがctf補正されたデータセットのようである。
注釈
本来のデータ解析の流れとの違い
スタック画像の取り込み、CTFデータ補正、データセットの編成は負染色データの場合、あまり意味が無いようにも思えるが このステップを順番に経ないと次の二次元クラス分類にいけない。
ヒント
画像が1枚だけの場合
画像が一枚だけの場合は、スタック画像の取り込み、CTFデータ補正、データセットの編成は省略していきなり 次の二次元クラス分類へ進める
二次元平均像の作成¶
いよいよ、二次元クラス分類を行う。
手順は、
を選択する。
注釈
の計算方法について
helixboxer.py の出力のような汚いスタック画像をそのまま使用する際には
ではなく
を使用する。
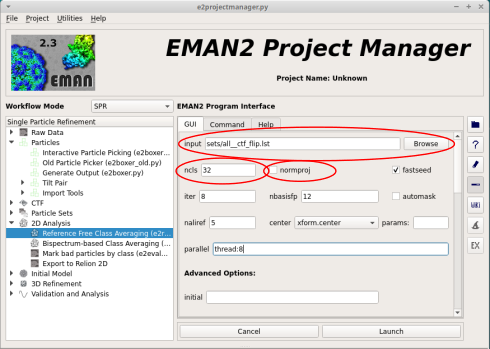
また、パラメータについても以下のように設定する。
- 入力値
input: | : sets/all__ctf_flip.lst |
|---|---|
ncls: | : 32 〜 20 |
nbasisfp: | : 12 など |
classaverager: | : mean |
parallel: | thread:8 |
他のオプション、 Launch のデフォルトは問題ないはずです
ヒント
二次元クラス分類について
このステップが最もエラーが出て途中でとまってしまう。
そのような場合は ncls を小さくしてみれば良い。ただし、あまり小さいと(例えば 10 未満)だと
三次元モデル作成がうまくいかない。
目安として一クラス平均の構成画像数が20以上500以下が推奨されている。
クラス平均の取捨選択¶
クラス平均のリファインメントをするために、
- 不良粒子を取り除く
- 良好な平均を選択する
を行い、データセットを再編成する。チュートリアルでのやり方と全く同じ。
- projectmanager.py 右上のフォルダアイコン
 のファイルブラウザーよりファイルを選択する。
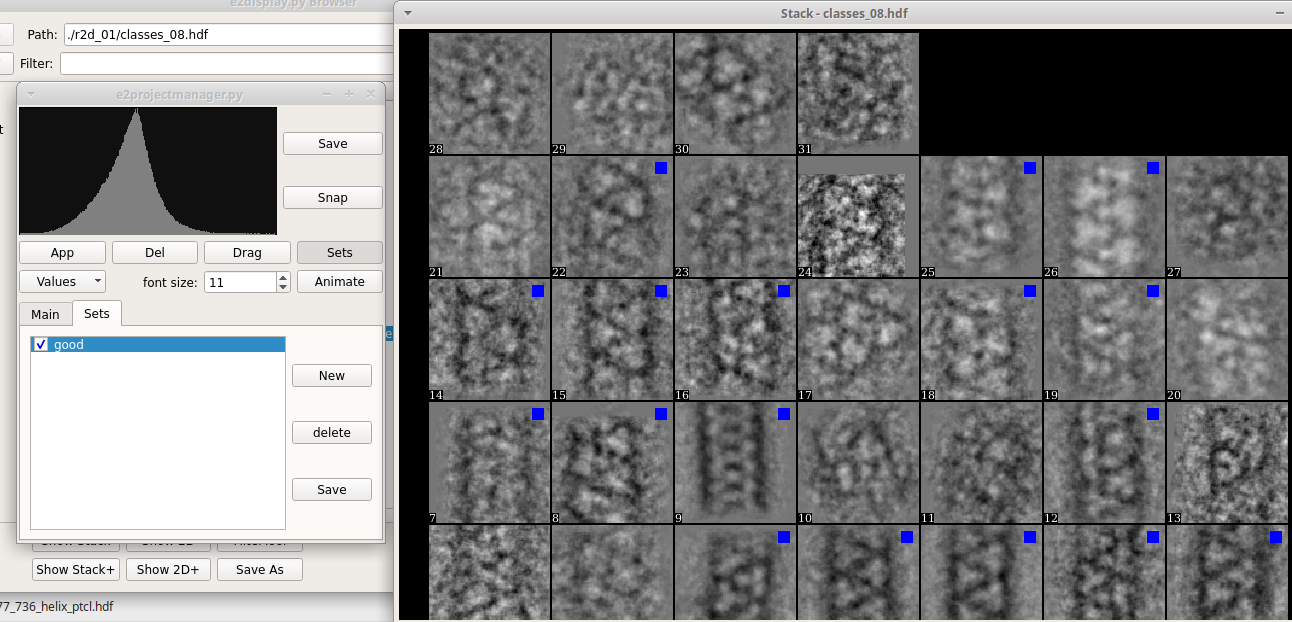
のファイルブラウザーよりファイルを選択する。 r2db_01を参照して、classes_08.hdfを選択する。(08はiterが8なため)- Show Stack+ をクリックするとクラス平均が表示される。
- クラス平均のウィンドウを中クリックすると新たなウィンドウが出るので、 sets ボタンを押してから、 sets タブをクリックする。
- New をクリックして
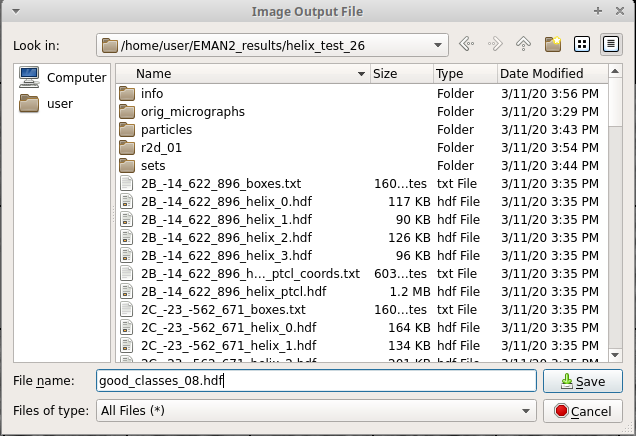
goodと入力する。また、 good をクリックしてアクティブにしておく。良いと思うクラス平均をクリックして選択する。選択すると青(もしくは緑)にマークされる。 - 右上の Save ボタンをクリックして
good_classes.hdfとして保存して、ウィンドウを閉じる。
スタック画像のクラス平均が出力される。その中の一つを中クリックすると新たなウィンドウ e2projectmanager.py が出る。
初期モデルの作成¶
クラス平均のリファインメントができたので、この二次元画像をもとに初期モデルを作成する。
手順は、
を選択する。
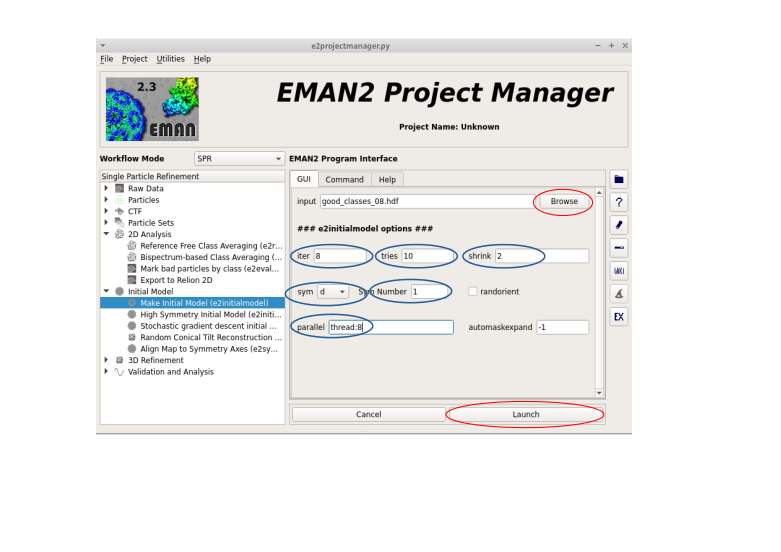
以下のパラメータを入力して、 Launch を押す。
- 入力値
input: | : good_classes.hdf |
|---|---|
sym Sym Number: | : d 1 |
iterations: | : 8 |
tries: | : 10 |
randorient: | : checkなし |
shrink: | : 2 |
parallel: | : thread:8 (PCのthreadの数) |
他のオプションにはデフォルト値を使用
注釈
このフィラメントの対称性は、2面回転対称なので軸対称性は D1
最適な初期モデルの選択¶
初期モデルの作成が終了すると、 initial_models というディレクトリーができるので最適な初期モデルの選択をする。
プロジェクトマネージャーウィンドウの右上のフォルダアイコンのファイルブラウザーよりファイルを選択する。
ファイルには、 model_NN_MM というファイルが確認できる。( N :プログラムの実行回数、 M :試行回数)
チュートリアルでは、10回の試行をしたので10個の初期モデルが生成している。 その中から、最適な初期モデルを以下の3つの観点から選ぶ。
- まず、
model_NN_MM.aptcle.hdfを確認する - 次に、
model_NN_MM.proj.hdfを確認する - 最後に、
model_NN_MM.hdfを確認する