チュートリアル¶
EMAN2 では、チュートリアルデータとしてβガラクトシダーゼのデータ
http://blake.bcm.edu/dl/workshop_2016_bgal.zip
が用意されているので、このデータを使ってソフトの使い方の学習することが可能。
このチュートリアルの目的としては、
- ソフトの使い方になれる
- 自分の計算機環境で解析ができるかの検証
として行う。
検証として実行する際には、チュートリアルデータとして用意された全てのデータを使うことは少々面倒である。 そこで、データの一部を抜粋して、解析ができるかを検証をした。
検証なので初期モデル作成までで最終的にモデルのリファインメントは行っていない。
使用したデータは、
BGal_000131,137,151,152,156 を使用した。(最低画像ファイルが5個あればテストはできる)
まず、解析手順の大まかな流れを以下に示す。
![// graphviz での作成の流れ
digraph G1 {
graph [size="6,4"];
node [shape=box];
a [label="EMAN2の起動"];
b [label="データの取り込み"];
c [label="電顕画像から粒子の抽出"];
d [label="データの補正"];
e [label="データセットの編成"];
e2 [label="",shape=circle];
a-> b [label="orig_micrographs/*.dm3"];
b-> c [label="micrographs/*.hdf"];
c-> d [label="particles/*.hdf"];
d -> e [label="particles/*.hdf"];
e -> e2 [dir=none];
{rank=same; a; e1;}
subgraph cluster1 {
label = "Modeling";
labelloc = "t";
labeljust = "l";
e1 [label="",shape=circle];
f [label="2Dクラス分類",shape=doubleoctagon];
g [label="クラス平均の取捨選択"];
h [label="初期モデル作成"];
f -> g [label="r2db#/classes*.hdf"];
g -> h [label="r2db#/good_classes.hdf"];
e1 -> f [label="sets/all_ctf_flip_lp5.lst"];
}
}](../_images/graphviz-5fe957d7ea61df1cc3a3695576440bad43d6a550.png)
図 2 EMAN2の概要¶
準備¶
チュートリアルデータのダウンロード¶
http://blake.bcm.edu/dl/workshop_2016_bgal.zip
よりチュートリアルデータをダウンロードする。
データは圧縮されているので、解凍する必要がある。
解凍方法は、ターミナルで以下のように入力して実行する。
$ unzip worshop_2016_bgal.zip
解凍すると、電顕画像と電顕画像上の粒子の座標情報が記録されている .box ファイルを確認することができる。
EMAN2 の起動¶
プロジェクトマネージャ e2projectmanager.py の起動¶
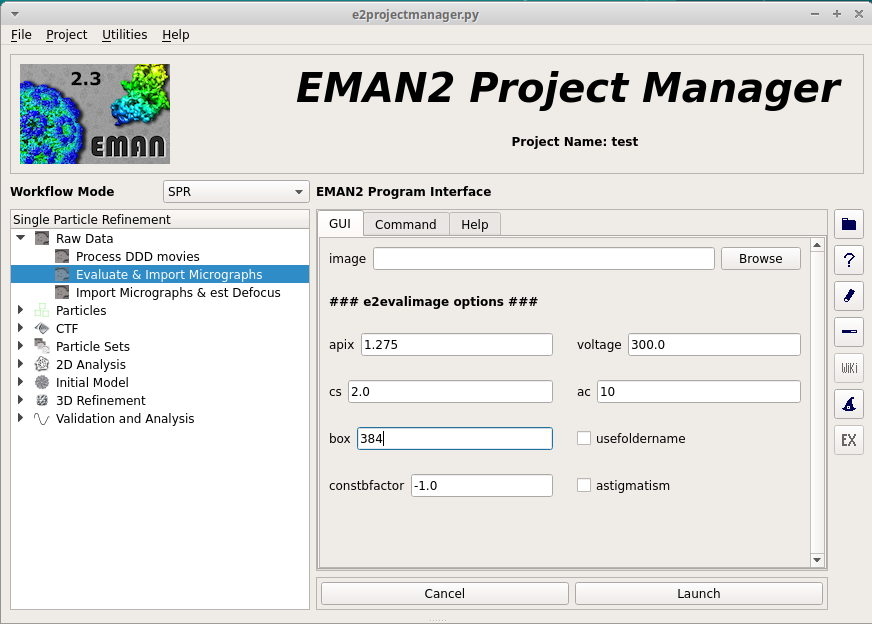
まず、 EMAN2 で電顕画像を処理するためにプロジェクトマネージャ e2projectmanager.py を起動する。
以下に示すように、解析する画像ファイルフォルダを含むプロジェクトディレクトリで、 e2projectmanager.py と入力して、実行する。
$ e2projectmanager.py
すると、プロジェクトマネージャが起動する。
パラメータの設定¶
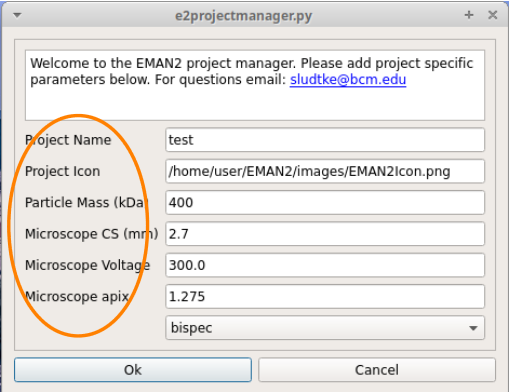
起動したら、はじめにプロジェクトのパラメータを設定する必要がある。
まず、 をクリック。
ウィンドウが表示されるので、パラメータを入力する。
チュートリアルでは、
| 質量: | = 400 |
|---|---|
| Cs: | = 2.7 |
| 電圧: | = 300 |
| apix: | = 1.275 |
を使用している。
ご用心
実験の際、パラメータのメモは忘れずに!
実際に解析をする際には、これらのパラメータを知っている必要がある。
これで、解析を行う準備ができたので、データの解析を進めていく。
データの取り込み¶
まず、プロジェクトにデータを取り込む。
データの取り込みに関しては、2つの方法がある。
- 一括ですべてのデータを取り込む
- 一枚ずつ確認して評価しながら適切と判断した電顕画像のみを取り込む
チュートリアルでは、比較的汚染の少ないきれいなデータを使用しているのですべてのフレームを一括で取り込む方法で問題はない。 電顕写真を評価して、適当なものだけを取り込むことも可能であり、自分のデータで解析する場合は、一つ一つデータを確認してからデータを取り込むほうが良い。
重要
ただ単にチュートリアルをなぞるだけなら最低5枚の画像があれば良い。 ファイル数が多くなると後の、自動粒子ピックアップの過程ですごく時間がかかる。
一括で取り込む場合¶
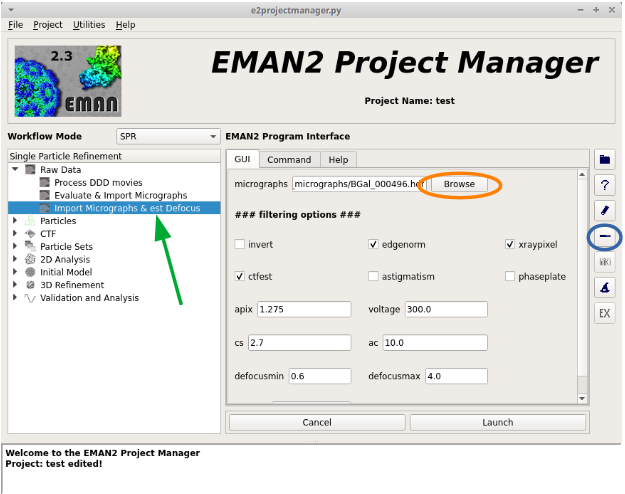
一括でデータを取り込む手順としては、
をクリック。
Browse をクリックして、 orig_micrographs のファイルを選択する。
パラメータ等の設定について、
edgenorm: | チェック入れる。 |
|---|---|
xraypixel: | チェック入れる。 |
ctfest: | チェック入れる。 |
invert: | チェック解除する。 |
以下のパラメータは、プロジェクトの編集をした際に入力されているので、後は Launch をクリックして実行する。
apixs |
1.275 |
voltage |
300 |
Cs |
2.7 |
ac |
10 |
defocus |
0.6-4.0 |
実行すると、画像がプロジェクトに取り込まれる。
選択して取り込む e2valimage.py¶
電顕画像を確認して、適切を思われるものを取捨選択して取り込む手順は、
をクリック。
Browse をクリックして、 orig_micrographs のファイルのすべての画像を選択する。
パラメータの設定は以下のように設定した。
apixs |
1.275 |
voltage |
300 |
Cs |
2.7 |
ac |
10 |
box |
384 |
constbfactor |
-1 |
box は、粒子サイズとは関係ない。
constbfactor は、デフォルトを使用した。
その他は、プロジェクトのパラメータである。
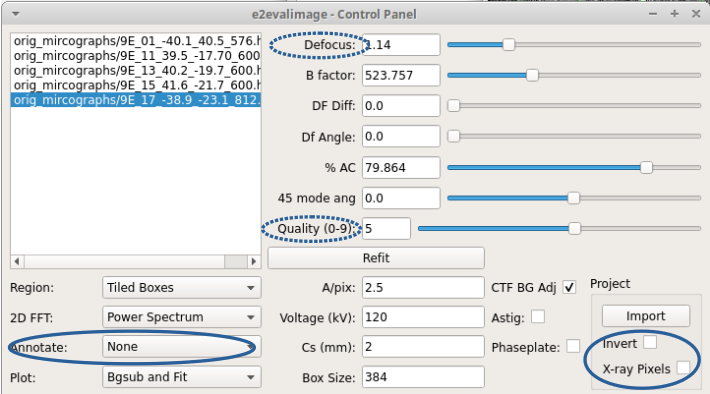
後は Launch をクリックして実行する。 実行すると、以下の4つのウィンドウが開く。
- コントロールパネル (Control Panel)
- 電顕画像ウィンドウ (e2valimage)
- View Plot (e2valimage-Plot)
- 2D FFT (e2valimage-2D FFT)
1. まず、コントロールパネルの Annotate の CTF Zeroes を None に変更する。
変更すると、2D FFT画面の線が消え干渉縞の様子が確認できる。
- また、
invertのチェックを解除する。
クライオデータでは、 X-ray Pixels はチェックを入ておく。
ご用心
負染色データの場合
X-ray Pixels: | unchecked |
|---|---|
% AC: | 80 |
解析対象の画像の区画
boxを指定する。- クリックすると
boxをOFFにできる。染色むらがある部分等は除く。 - 除いた部分は、 View Plot での計算に使われない。
- クリックすると
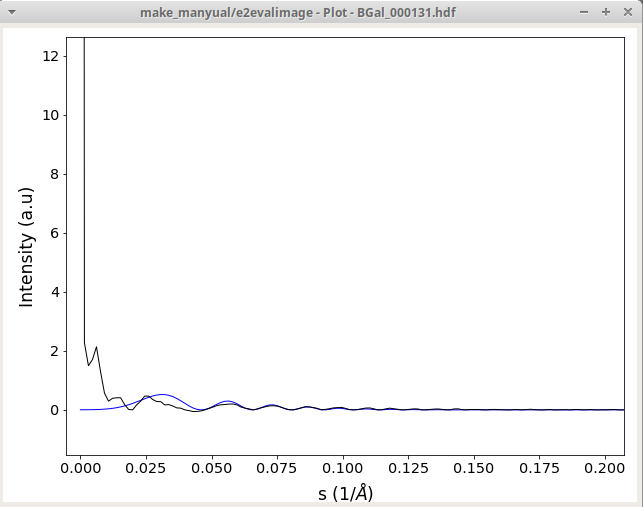
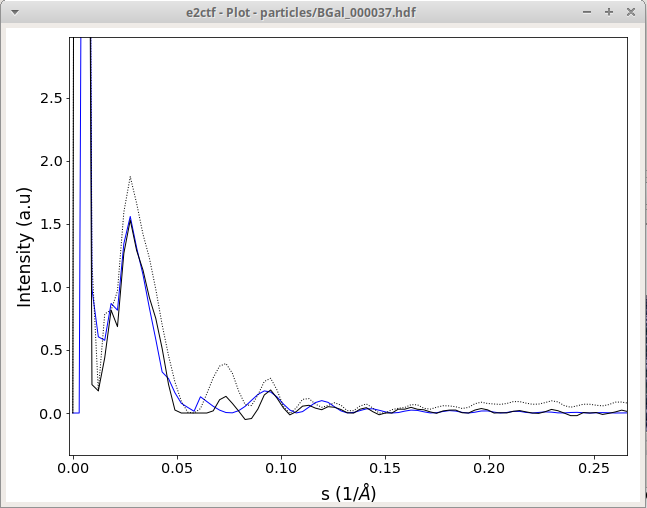
View Plot には、円環平均され、バックグラウンドを差し引いたパワースペクトルが表示されます。
右ドラッグしてズームして干渉線が見えるようにしておくこと。
黒はバックグラウンド、青はパラメータより計算された曲線。
Defocusに関してはCTF補正のところで詳説する。
警告
box の ON/OFF について
box で囲まれた領域では、 ON/OFF が実際に切り替わっているか確認はとれていないので、処理を行う際には、結果に注意。
- 実際の画像選択
以下の3つの点に注意して取り込む画像を選択していく。
- ドリフト
- 乱視
- 粒子濃度
ここで、ドリフトとは、FFTの干渉縞に以下のように方向性減衰が見られることである。 乱視とは、FFTが楕円になってしまっている。
また、粒子濃度とは、電顕画像で粒子間が接近しすぎている場合のこと。
これらの場合は取り除くか、低い品質値として取り込む。
Quallity を指定することで、後の工程で低クオリティーの電顕画像を取り除くことも可能。
画像選択の操作方法のショートカットについては、
この作業以降では、次を使用できます。
- 上下矢印 -次/前の画像を選択
- 左/右矢印 -ピンぼけの小さな変化
- i -顕微鏡写真をプロジェクトにインポートします
- u -誤ってインポートした顕微鏡写真のインポートを解除します
- 0-9 -品質を設定します(インポート前)、(5がデフォルト値)
注釈
不良データの除きもれについて
不良データを除くステップは後の工程でもある。また、不良データを取り除きすぎるとモデルの偏りにもつながりモデルの再構築の品質を低下させることにつながる。 そのため大まかでも大丈夫そうである。
- 適当な電顕画像を取り込んだら、コントロールパネルを閉じて終了する。
注釈
e2evalimage.py の機能について
e2evalimage.py はさらに多くの機能があり、記載したのは使い方の一例。
詳しくは https://blake.bcm.edu/emanwiki/EMAN2/Programs/e2evalimage 参照のこと。
電顕写真から粒子の抽出 e2boxer.py¶
次に、電顕画像から粒子を抽出します。
チュートリアルでは、すでに粒子の座標情報が記録された .box ファイルが用意されている。
しかし、通常は e2boxer.py を用いて粒子の抽出を行う必要がある。
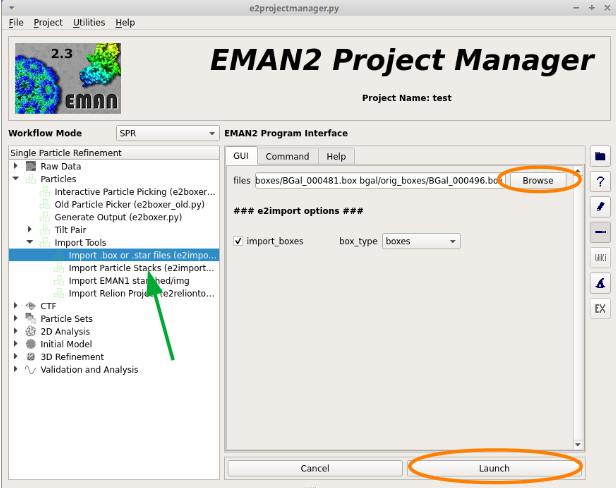
.box ファイルより粒子の抽出¶
チュートリアルでは、粒子の座標情報が記録されている .box ファイルより粒子の抽出を行う。
粒子の抽出手順は、
をクリック。
ファイル、パラメータを指定して、実行する。
ご用心
チュートリアルとの違い
チュートリアルでは、粒子の座標情報が記録された .box ファイルが用意されている。
自分でデータを解析する際は、粒子を抽出する必要がある。
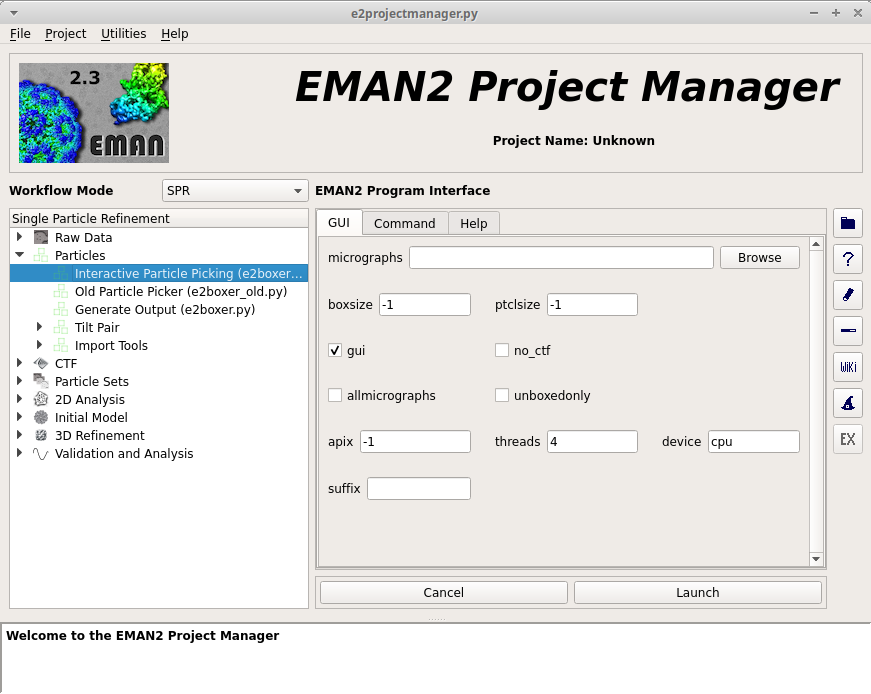
e2boxer.py による粒子の抽出¶
チュートリアルでは、すでに粒子が抽出されていて抽出した粒子の座標情報が記録された .box ファイルより粒子の抽出ができる。
しかし、通常は、電顕画像から粒子を抽出する必要がある。
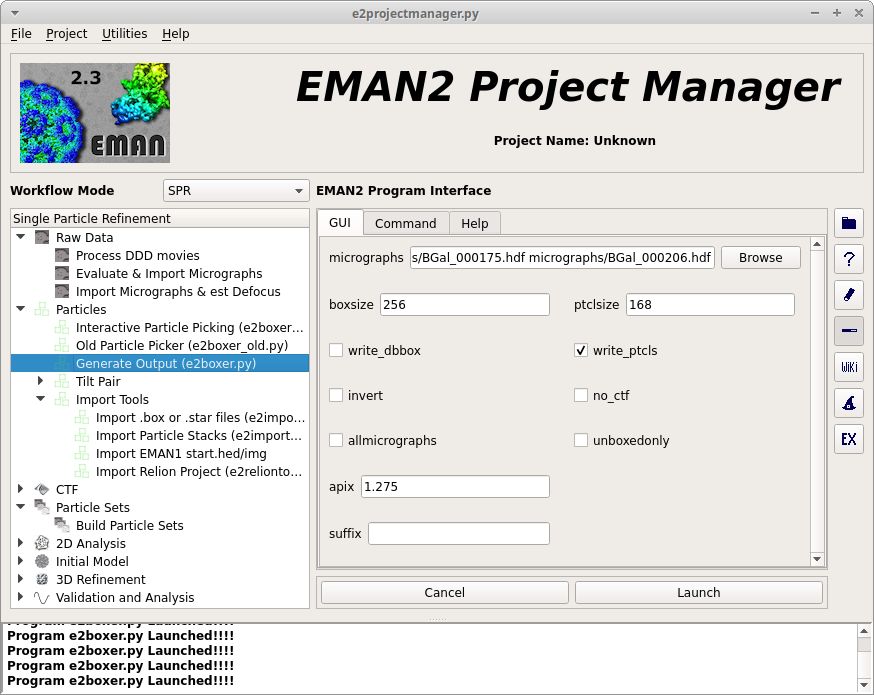
粒子の抽出するための操作は、
をクリック。
必要なパラメータを設定する。
| boxsize: | =256 |
|---|---|
| ptclesize: | =168 |
とした。ここで粒子の最大軸の長さが168*1.27=214 Aぐらいになっていることに注意。
注釈
boxsize と ptclesize について
抽出する粒子サイズに応じて、 ptclesize を設定する必要がある。 boxsize は 粒子サイズの最大軸の1.5-2倍程度の数値にしなければいけない。
これらの適当な数値については、任意の値ではなく以下のセットを参照する。
16, 24, 32, 36, 40, 44, 48, 52, 56, 60, 64, 72, 84, 96, 100, 104,
112, 120, 128, 132, 140, 168, 180, 192, 196, 208, 216, 220, 224,
240, 256, 260, 288, 300,320, 352, 360, 384, 416, 440, 448, 480,
512, 540, 560, 576, 588, 600, 630, 640, 648, 672, 686, 700, 720,
750, 756, 768, 784, 800, 810, 840, 864, 882, 896,900, 960, 972,
980, 1000, 1008, 1024
後は Launch をクリックして e2boxer.py を実行する。
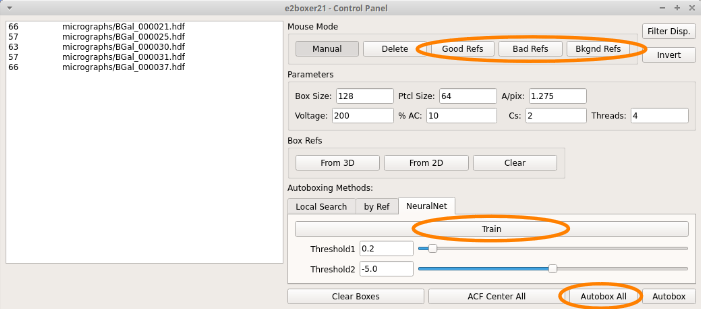
実行すると、コントロールパネル、電顕画像、粒子ウィンドウが開く。
- コントロールパネル Good Refs を選択して、電顕画像より良いと思われる粒子像を参照する。(最低5つは必要)
- コントロールパネル Bkgnd Refs を選択して、電顕画像よりバックグラウンドと思われる部分を参照する。(最低5つは必要)
- 粒子像とバックグラウンドを参照したら、 をクリックして、参照した粒子像、バックグラウンドより特徴抽出を行う。
- ターミナルに
Training ...と表示されその次にiteration 0, cost -**と表示されていれば深層学習が進行中のはず。かなり時間がかかるはず。 - ターミナルを確認してトレーニングが終了したら、 Autobox All をクリックして電顕画像より粒子を自動抽出を行う。
注釈
programの動作状況について
Task Manager ウインドウで何も表示されていない状態ならプログラムは終了している。
ご用心
e2boxer.py について
このプログラムは深層学習により選別する粒子像、バックグラウンドを学習する。従って、あらかじめ tensorflow-gpu がインストールされていないとエラーが出る。
注釈
訓練データの数について
参照するバックグラウンドのは、最低5つ必要だが多いほうが望ましい。
また、必要に応じて Bad Ref も指定する。
自動抽出を行う際は、しきい値はデフォルトを使用したが、変更することで変化があるかもしれない。 しきい値はデフォルトで行った。
その他、いろいろノウハウはあるみたい。
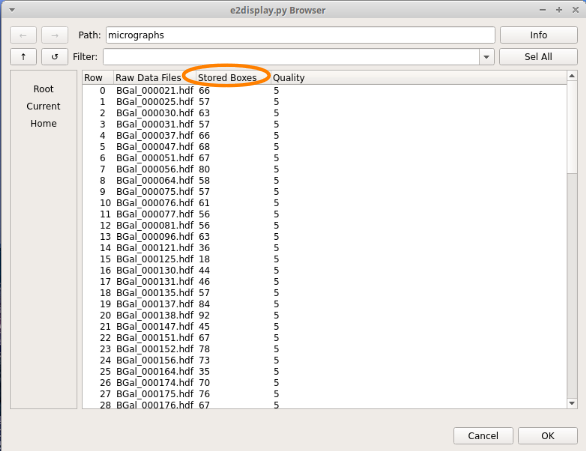
結果の確認¶
抽出が完了したので、
より結果の確認を行う。
実行した結果、 Stored Boxes の値が、 0 でなければOK
ヒント
Stored Boxes の値について
数値が 0 だった場合は、粒子の抽出がうまくできていないので、再度粒子の抽出を行ってみる。
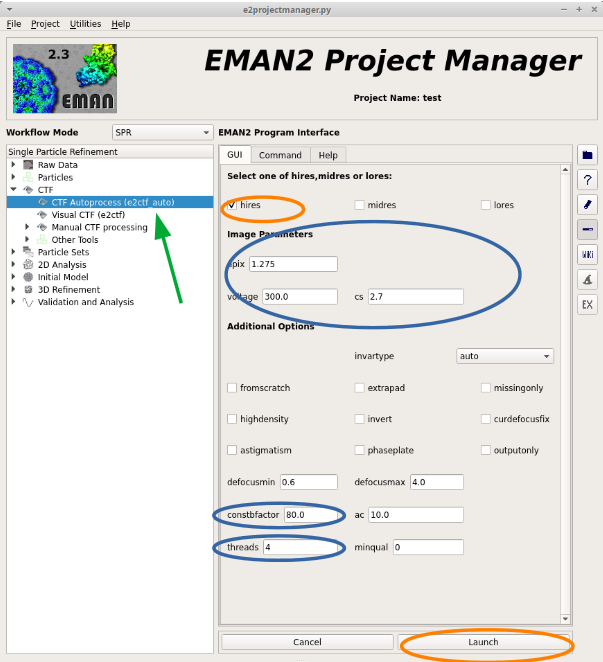
データの補正 e2ctf_auto.py¶
データの補正では、
- CTFパラメータ
- 位相反転
- 振幅補正
を行う。
また、補正に関しては自動で行われる。
操作手順は、
を選択する。
hires にチェックを入れて、その他はデフォルトで実行する。
他に、 constbfactor を変更するほうがいいのかも。例えば 80 とか。
ご用心
負染色データの場合
hires: | unchecked |
|---|---|
lores: | checked |
補正の確認 e2ctf.py¶
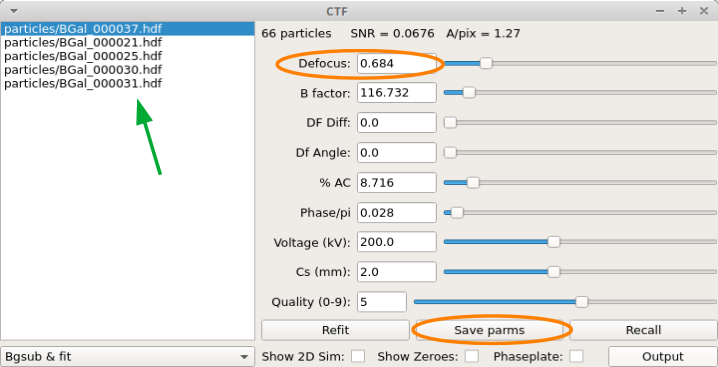
オートフィットは、精度良く実行される。しかし、まれにフィッティングエラーが起こることがある。 そのため、フィッティングエラーがないことを確認する。
自動補正ができていない場合は、コントロールパネルの Defocus の値が大きく異なって出力される。
なので、 Defocus の値を再調整して、 Save params で保存する。
調整は、プロットウィンドウの黒と青の曲線がピークが合うように調整する。 また、黒はバックグラウンド、青はパラメータより計算された曲線。本来であればコレが一致する。
ヒント
データ補正について
補正の確認は、チュートリアルでは、必要はない。
自分でデータを解析する際は、確認したほうが良い。
この段階では青線と黒線が完璧に一致している必要はない。大切なのはゼロ点の位置。
データセットの編成 e2buildsets.py¶
CTFの補正を行い終了するとパーティクルデータセット( .lst ファイル)を含んだ sets ディレクトリが生成し、
三次元初期モデルはそのデータセットより構築される。
このステップで、データセットの不良粒子を除いたり、データを取り込む際に、低品質値に割り当てた像を除いたりすることで編成(データの取捨選択)を行う。 様々な編成のデータセットを作成し、それにより、三次元モデリングの結果が変わり検討していく。
チュートリアルでは、最初は、すべての粒子を含んだ状態で、 all と名前をつけてパーティクルデータセット( all.lst )を作成している。
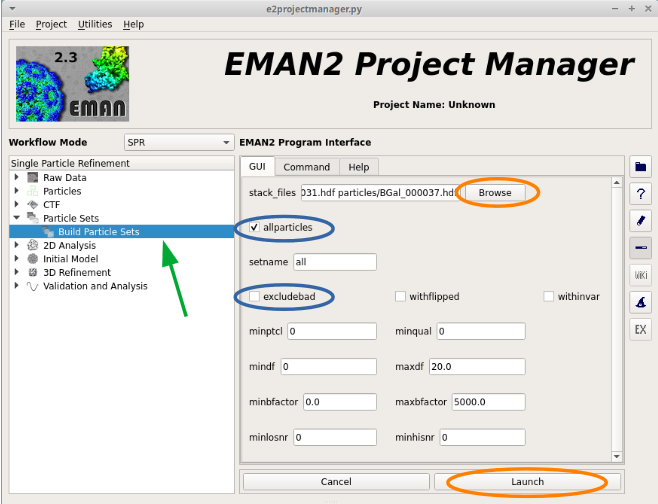
を選択する。
- stack_files で
particlesのディレクトリにあるスタック画像を選択する。allparticlesをチェックし、excludebadチェックボックスを外す。- セット名ボックスに任意の名前を入力する。ここでは、「
all」と入力- 顕微鏡写真を手動で評価するときに低い品質値を指定しといた場合、
minqualボックスに5を入力することもできます。これにより、品質が5未満に設定された画像が除外できる- Launch を押す。
ヒント
データセットと三次元モデル構築
三次元モデルは、複数のCTFデータ補正された粒子のスタック画像(パーティクルデータセット)( .lst ファイル)から作成される。
このステップで、様々なパーティクルデータセット( .lst ファイル)を作成することで条件の異なったパーティクルセットより三次元モデルの構築ができる。
3次元モデリング¶
パーティクルデータセット( .lst ファイル)をもとに、三次元モデリングをしていく。
三次元モデルを構築するにあたり、いくつかのステップがある。
流れの概要は、
- 二次元クラス平均を生成
- クラス平均の取捨選択
- 初期モデルの作成
- 三次元モデルの精密化
である。
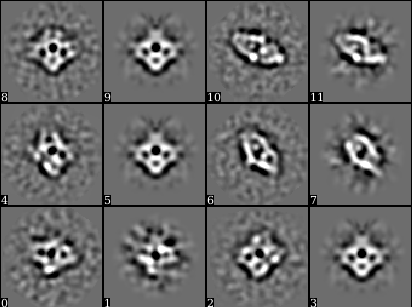
クラス平均の生成 e2refine2d_bispec.py¶
まずは、クラス平均の作成を行う。
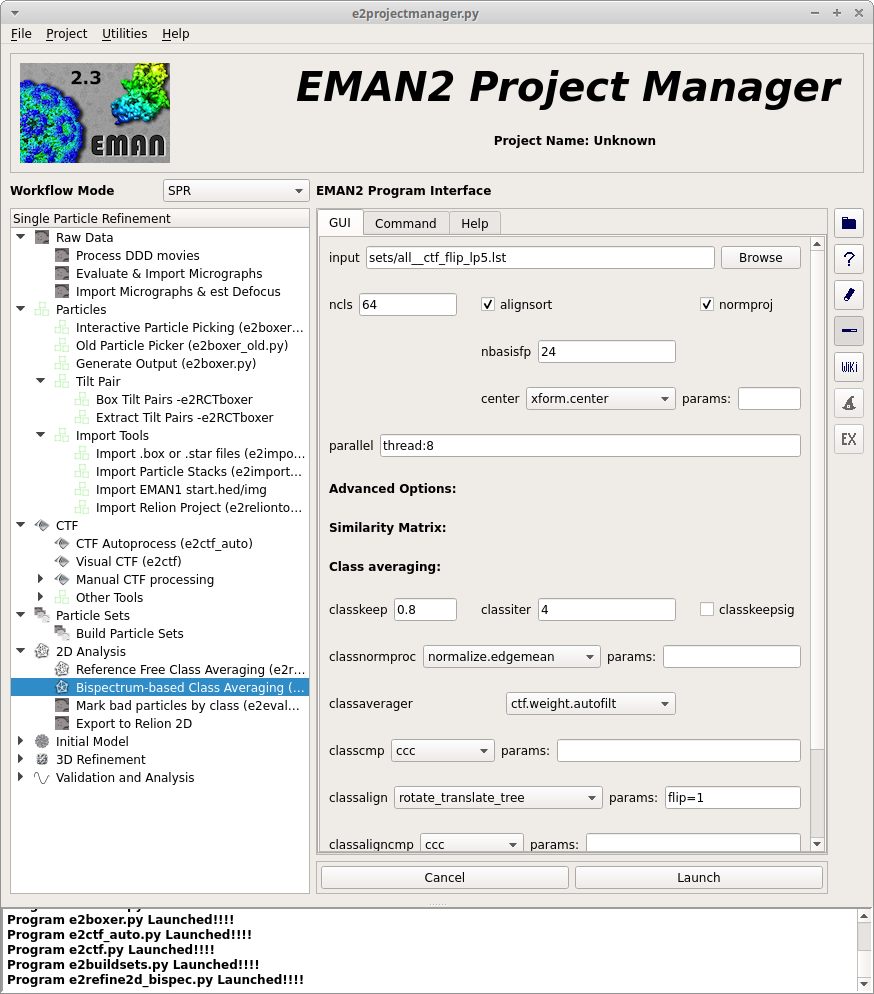
手順は、
を選択する。
また、パラメータについても以下のように設定する。
- 入力値
input: | : sets / all__ctf_flip_lp5.lst |
|---|---|
Ncls: | : 64 |
nbasisfp: | : 24 |
classalign: | : rotate_translate_tree |
params: | : flip=1 |
parallel: | : thread:4 (またはコアの数) |
classaverager: | : ctf.weight.autofilt |
その他はデフォルト値
ヒント
クラス平均のオプションについて
norproj のチェックボックスはどうするべきかの検討をする必要があるかも。
その他、オプションについても検討が必要かもしれない。
クラス平均の取捨選択¶
クラス平均の取捨選択では、
- 不良粒子を取り除く
- 良好な平均を選択する
を行う。
不良粒子の一部を除去 e2valparticles.py¶
後半のステップでも、不良粒子を自動的に除去するステップがあるため、この操作はオプションである。 しかし、完全には取り除くことができない場合もあることには注意が必要。
そのため、クラス平均を生成したら、不良粒子が存在するクラスがあるがを確認する。
- を選択する。
- Launch を押す。
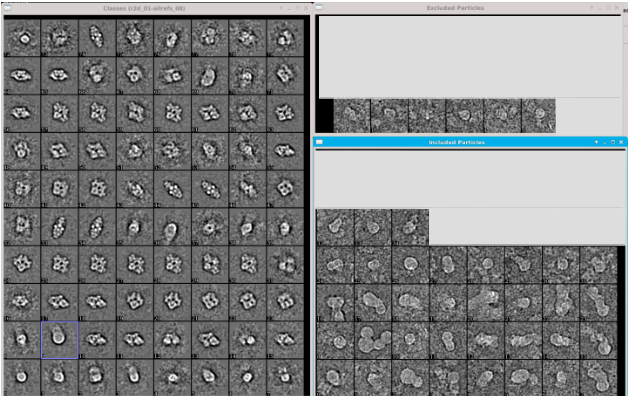
ウィンドウ1が開く。
ウィンドウ1が開いたら、r2bd_01/classes_00.hdfを選択する。 オプションはデフォルトすると、新たに3つのウインドウ
ウィンドウ2が開く。Classeswindowでクラス平均を確認する。そして、不良粒子で構成されているクラス平均をダブルクリックすると、右上に青のマークが表示される。
不良粒子が含まれるクラス平均をマークしたら、 Mark as Bad をクリック。
Are you sure ?とやり直しはできないとウインドウが開き尋ねられるが、ここはOKで次に進む。完了したら
ウィンドウ1を閉じて、 e2valparticles.py を終了させる。
ヒント
良い粒子の寄与と悪い粒子の寄与
良い粒子の寄与と悪い粒子の寄与では、 悪い粒子 の寄与の方が結果に大きく影響を与える。
なので、良い粒子を除くことになっても不良粒子を取り除くことのほうが重要。
また、80-90%の不良粒子を取り除ければOK。全ては無理。
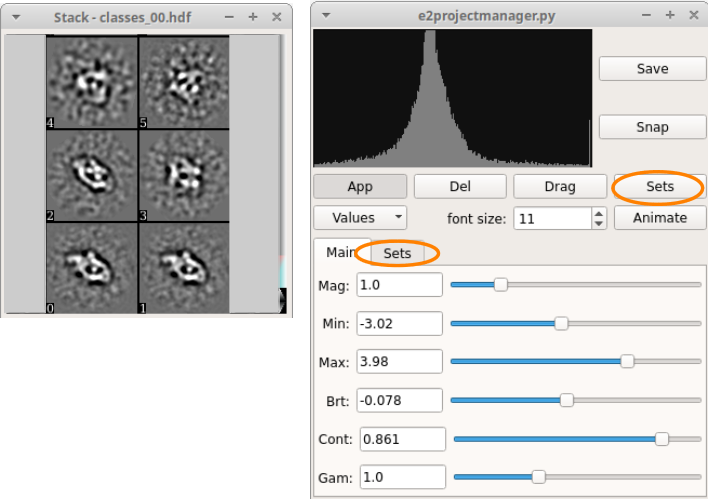
良好なクラス平均の選択¶
- プロジェクトマネージャーウィンドウの右上のフォルダアイコンのファイルブラウザーよりファイルを選択する。
r2db_01を参照して、classes_00.hdfを選択する。- Show Stack+ をクリックするとクラス平均が表示される。
- クラス平均のウィンドウを中クリックすると新たなウィンドウが出るので、 sets ボタンを押してから、 sets タブをクリックする。
- New をクリックして
goodと入力する。また、 good をクリックしてアクティブにしておく。良いと思うクラス平均をクリックして選択する。選択すると青(もしくは緑)にマークされる。 - 選択したら、
good_classes.hdfとして保存して、ウィンドウを閉じる。
ご用心
ver2.31とver2.22 のtutorialの違い
ver2.31とは、操作手順が微妙に異なる。
選択しているファイルが、 classes_00.hdf でななく classes_sort_00.hdf である。
また、ver2.31では、いいものを選択しているが、ver2.22では悪いものを除いている。
どちらが良いのだろうか??
注釈
焦るな
たまに、この過程でコアダンプで落ちてしまった。
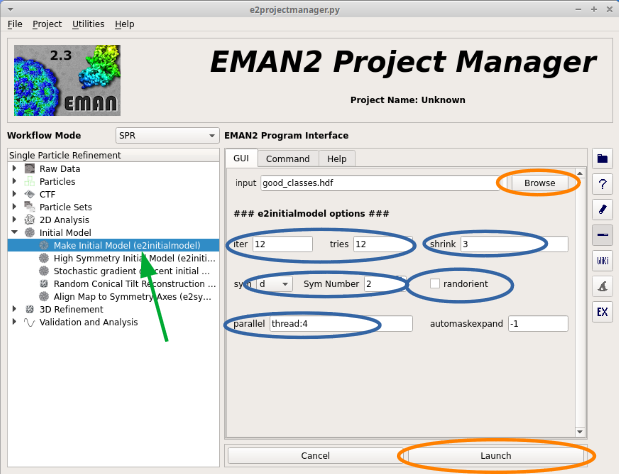
初期モデルの作成 e2initialmodel.py¶
クラス平均の取捨選択ができたので、この二次元画像セット good_classes.hdf をもとに初期モデルを作成する。
手順は、
を選択する。
以下のパラメータを入力して、 [Launch] を押す。
- 入力値
input: | : good_classes.hdf |
|---|---|
sym: | : D2 |
iterations: | : 12 |
tries: | : 12 |
randorient: | : checkなし |
shrink: | : 3 |
parallel: | : thread:4 (PCのthreadの数) |
他のオプションにはデフォルト値を使用
実行結果を参照して、最適な初期モデルを選択する。
ご用心
実行が終了するまでかなり時間がかかる
最適な初期モデルの選択¶
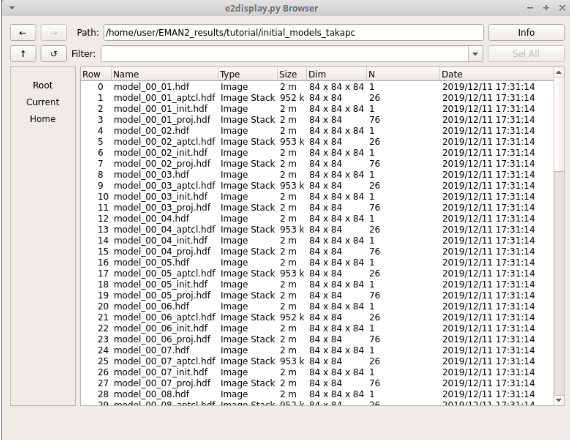
初期モデルの作成が終了すると、 initial_models というディレクトリーができるので最適な初期モデルの選択をする。
プロジェクトマネージャーウィンドウの右上のフォルダアイコンのファイルブラウザーよりファイルを選択する。
ファイルには、 model_NN_MM というファイルが確認できる。( N :プログラムの実行回数、 M :試行回数)
チュートリアルでは、12回の試行をしたので12個の初期モデルが生成している。 その中から、最適な初期モデルを以下の3つの観点から選ぶ。
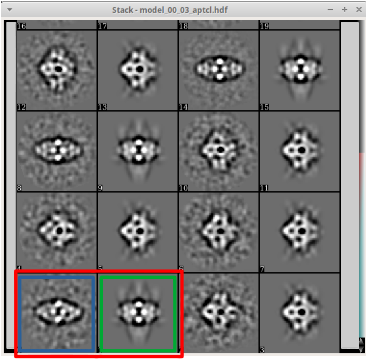
1. まず、 model_NN_MM.aptcle.hdf を確認する¶
model_NN_MM.aptcle.hdf ファイルを選択し、 Show Stack+ をクリックすると、
model_NN_MM.aptcle.hdf ファイルの中身は、2つの画像のペア(赤)がいくつか表示される。
青は、生成したクラス平均。緑は、初期モデルの投影像。
適切な初期モデルであればこれらがよく一致する。
いい例では、どれも平均像と投影像が一致しているが、悪い例では、あまり一致しているようには見えない。
ヒント
良い例と悪い例の見分け方
例としては、チュートリアルのマニュアルを参照すると良い。 https://blake.bcm.edu/emanwiki/EMAN2/Tutorials?action=AttachFile&do=view&target=EMAN2_SingleParticleTutorial.pdf
このような失敗の原因としては、
- 初期モデルが良くない
- クラス平均が良くない
- 粒子母集団が不均一
であることが考えられる。そのため、再度実行する。試行回数を増やすなどする。
2. 次に、 model_NN_MM.proj.hdf を確認する¶
model_NN_MM.proj.hdf ファイルを選択し、 Show Stack+ をクリックすると、
様々な方向からの投影像が表示されている。
悪い初期モデルでは、歪んで見える。
悪い例では、投影像の多くは歪んでおり。B-ガラクトシダーゼの投影であるようには見えないものが多い。 いい例も十分ではないが、歪んでいる投影像は少なく、B-ガラクトシダーゼの投影らしく見える。
3. 最後に、 model_NN_MM.hdf を確認する¶
適切な3次元モデルができているかを確認する。
model_NN_MM.hdf ファイルを選択し、 Show 3D をクリックすると、
三次元モデルが表示される。
これらの確認により、最適な初期モデルの一つを特定する。
ひとまず、三次元初期モデルを構築できた。