1. PISA の概要¶
1.1. PISA とは¶
- PDBePISA (Proteins, Interfaces, Structures and Assemblies) http://www.ebi.ac.uk/pdbe/pisa/ :
- PDBe(Protein Data Bank Europe)によって提供される、PDBファイル中の高分子複合体(タンパク質、DNA、RNA、リガンドなど)の 構造安定性を調べるためのWebサービスである。 タンパク質の界面の構造を調べたり、四次構造を予測したり、類似性した相互作用を持つ構造体を検索したりすることができる。
1.1.1. PISA の解析対象¶
PISA には解析対象として2種類ある
| Structure Analysis: | |
|---|---|
| PDBにエントリーされている結晶構造や、PDBファイル形式のモデル構造。 通常の対象は複数の鎖を含むPDBファイルであるがモノマーのPDBファイルでも良い。 PDBのエントリー番号を指定したり、PDBファイルをアップロードする。 | |
| Database Searches: | |
| PDBのデータベースを対象として条件にある四次構造を持つ構造体を検索する。 | |
1.1.2. PISA でできること¶
PISA には3つの解析モードがある。
interfaces: | サブユニット間、およびサブユニットと基質の界面 |
|---|---|
Monomers: | 複合体を構成する各サブユニットの溶媒との界面 |
Assembles: | 複合体中のサブユニット界面 |
1.2. 理論的背景¶
PISA における解析の対象はPDB形式の原子座標ファイルである。 PDBファイルには生物学的集合体がモノマーでもオリゴマーでも良いが、 通常は複数のポリペプチド鎖からなるpdbファイルである。
PISA で扱う結晶中でのタンパク質の界面
- 隣接する単位格子の分子との界面
- 隣接する非結晶構造単位間での界面
PISA における、タンパク質の構造安定性は以下の物理化学的性質によって決定される。
- 会合の自由エネルギー
- 溶媒和エネルギーの獲得
- 界面の面積
- 水素結合
- 界面に働く塩橋
- 疎水性特異部位
PISA における四次構造の予測は単位格子にとどまらない。
- 単位格子をまたいだ四次構造の予測
- 「結晶中での相互作用が溶液中でも起きているから結晶ができる」と推測し、PISA が上記の六つの基準で複合体を予測している。
2. 操作方法と結果の見方¶
2.1. PISA の起動¶
PISA のサイト(http://www.ebi.ac.uk/pdbe/pisa/)を開き、 Launch PDBePisa をクリックする。
最初に Structure Analysis モードの説明を行う
2.2. Structure Analysis による解析¶
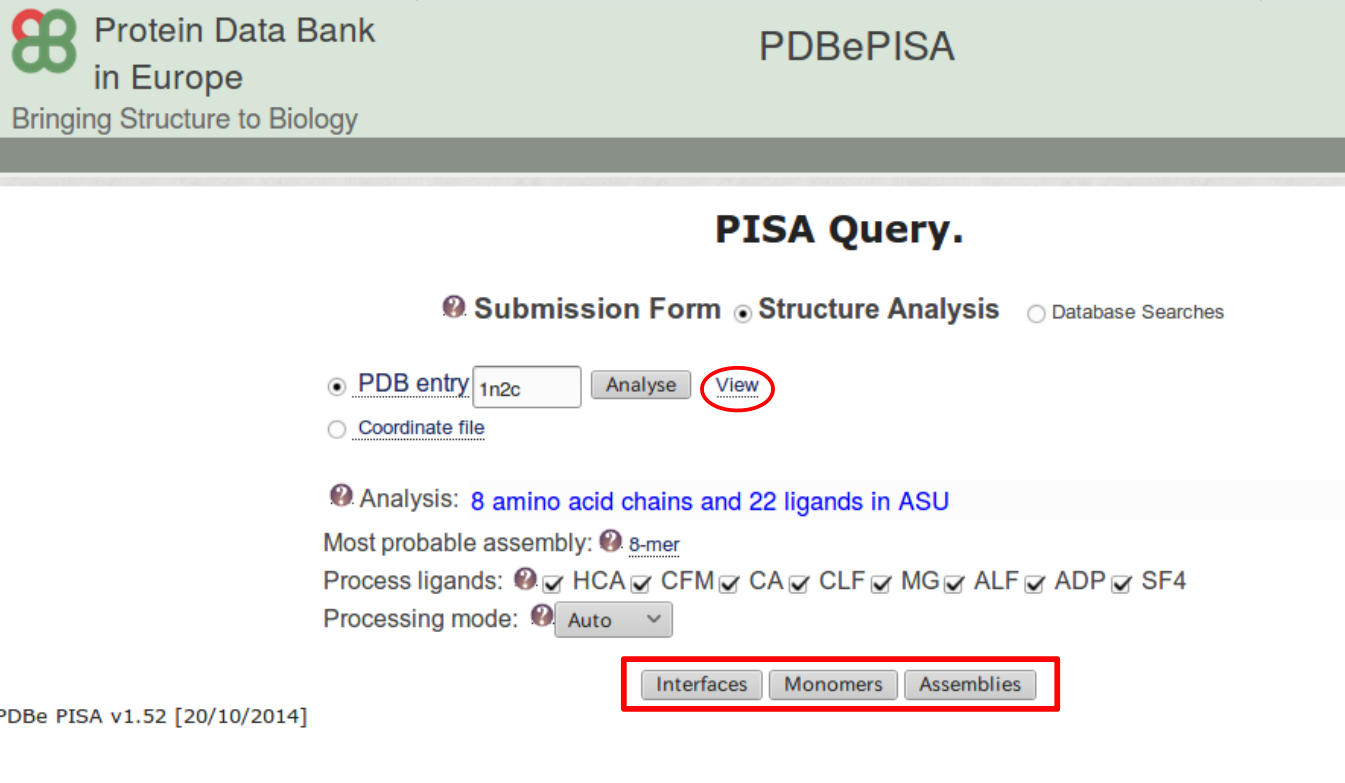
図 2.2 で調べたいPDBエントリー名を入力する。
図 2.3 ではタンパク質鎖の数と結合リガンドの数などPDBエントリーに関する予備情報が得られる。 また必要なリガンドを選択したり処理モードを選択したりする。 図 2.3 で view をクリックすると構造を表示することができる。
図 2.3 において Interfaces 、 Monomers 、 Assebblies をクリックすると目的のタンパク質の構造的性質を知ることができる。
2.2.1. Monomers¶
一つ一つのタンパク質鎖やリガンドに関する以下のような情報が得られる。
解析リスト中の項目とその内容を下の表にまとめた。
| 項目 | 内容 |
|---|---|
Nat |
全ての原子の数 |
Nres |
全ての残基の数 |
sNat |
表面の原子の数 |
sNres |
表面の残基の数 |
Area |
溶媒分子に接触している面積 |
| \(\Delta G\) | 溶媒和自由エネルギー |
1n2c の場合では A〜H のタンパク質鎖と
HCA , CFM , CA , CLF , MG , ALF , ADP , SF4 というリガンドに関する情報が得られる。
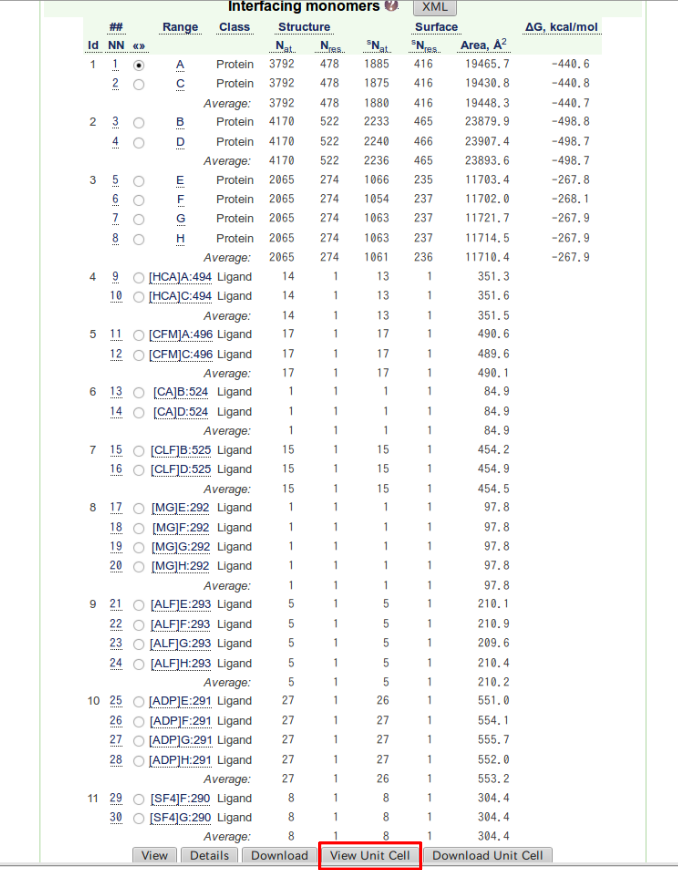
図 2.5 Monomers の解析結果
id がふられている。
NN をクリックするとタンパク質鎖中のどのアミノ酸が溶媒分子と接しているか、などがさらに詳しく分かる。1n2c の場合、 A , C 鎖、 B , D 鎖、 E 〜 H 鎖が
それぞれ同一の分子であるのでそれぞれひとまとめに分類されている。
また、 View Unit Cell で 1n2c の単位格子を表示することができる。
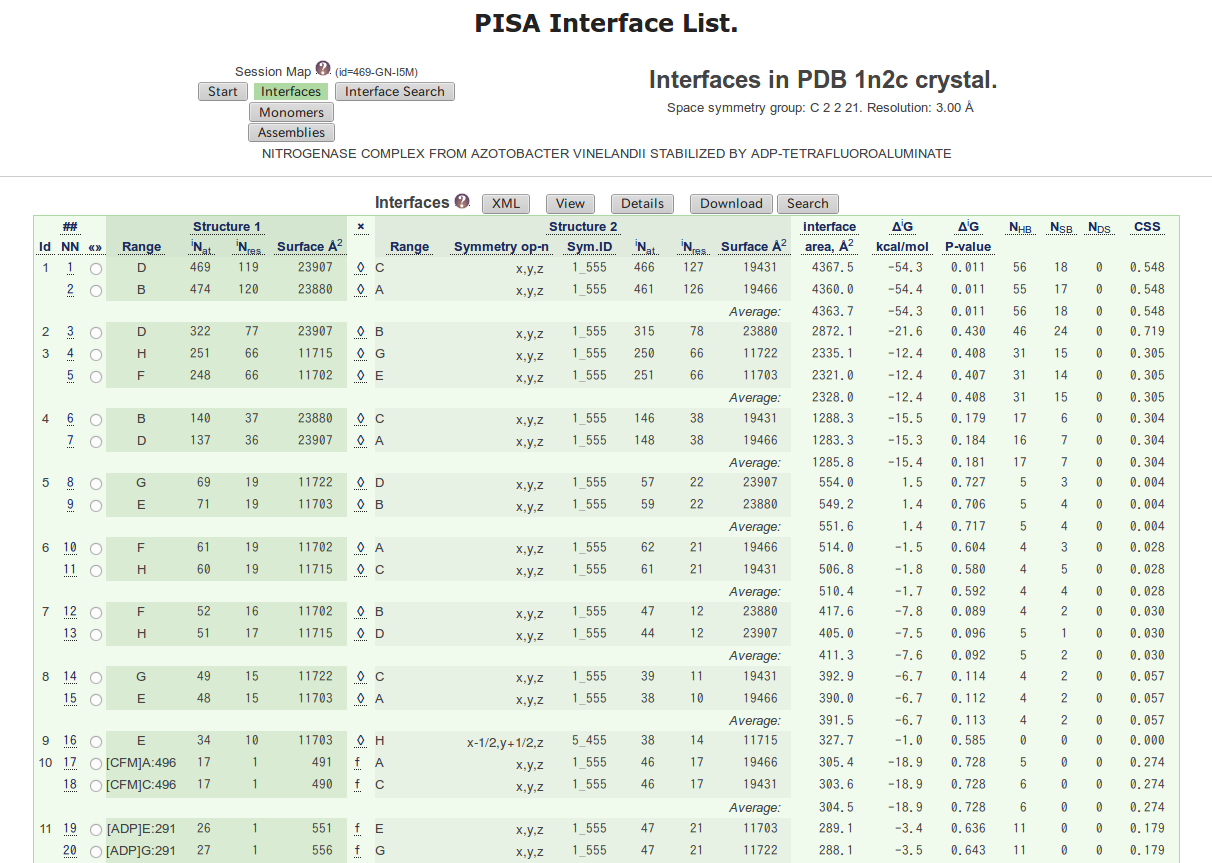
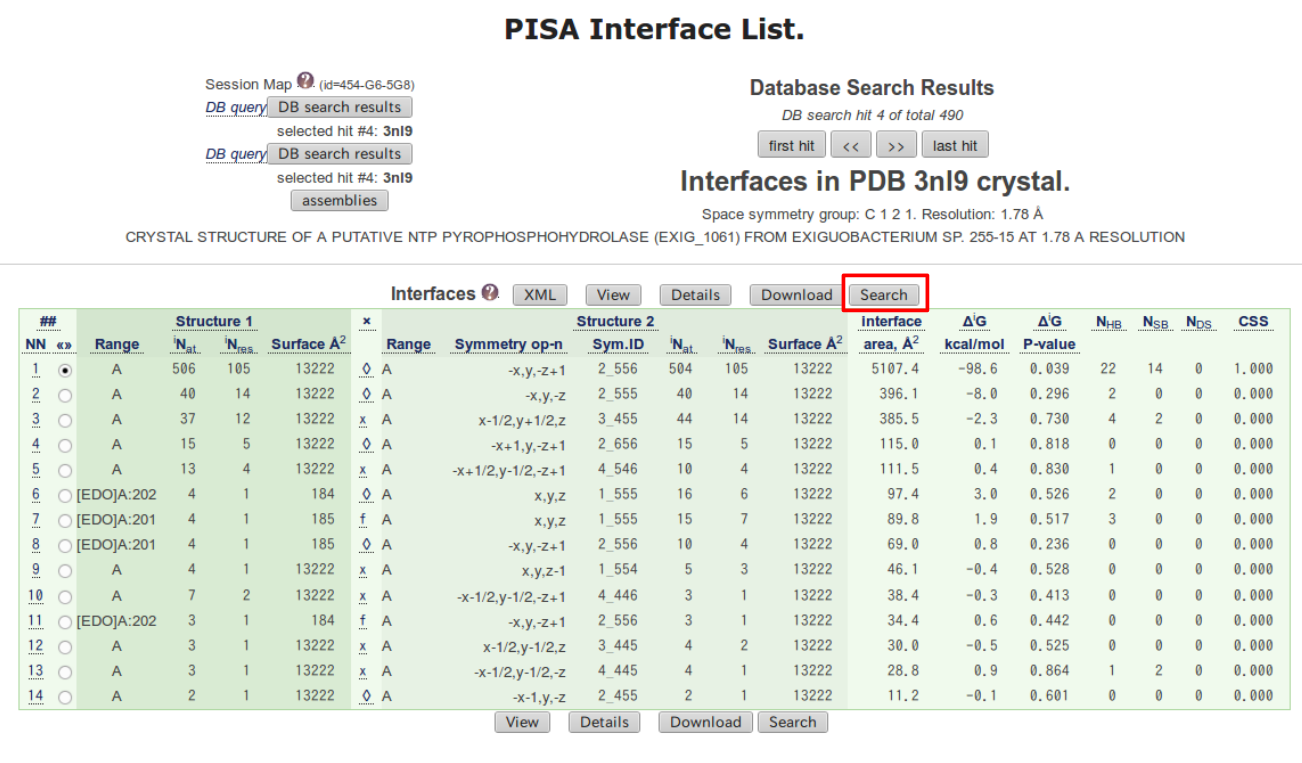
2.2.2. Interfaces¶
2つのタンパク質鎖間やタンパク質鎖と低分子間の界面に関する以下のような情報が得られる。
リスト中の項目とその内容を下の表にまとめた。
| 項目 | 内容 |
|---|---|
iNat |
界面として接する原子の数 |
iNres |
界面として接する残基の数 |
Symmetry op-n |
対称操作 |
Sym.ID |
単位格子の位置 |
Interface area |
界面の表面積 |
| \(\Delta_iG\) | 界面形成時の溶媒和エネルギーの変化量 |
| \(\Delta_iG\, \rm{P-value}\) | 相互作用のしやすさを表す。
P<0.5であれば相互作用しやすくP>0.5であれば相互作用しにくい。
|
N(HB) |
界面に働く水素結合の数 |
N(SB) |
界面に働く塩橋の数 |
N(DS) |
界面に働くジスルフィド結合の数 |
CSS |
界面が集合体の形成にどの程度重要であるかを表すスコア |
図 2.7 で x の列の \(\diamondsuit\) をクリックすると構造上どの位置の界面か確認することができる。
図 2.7 で NN をクリックすると、どのアミノ酸残基に結合が生じているかや
どのアミノ酸が界面を形成しているかなどの詳しい情報が得られる。
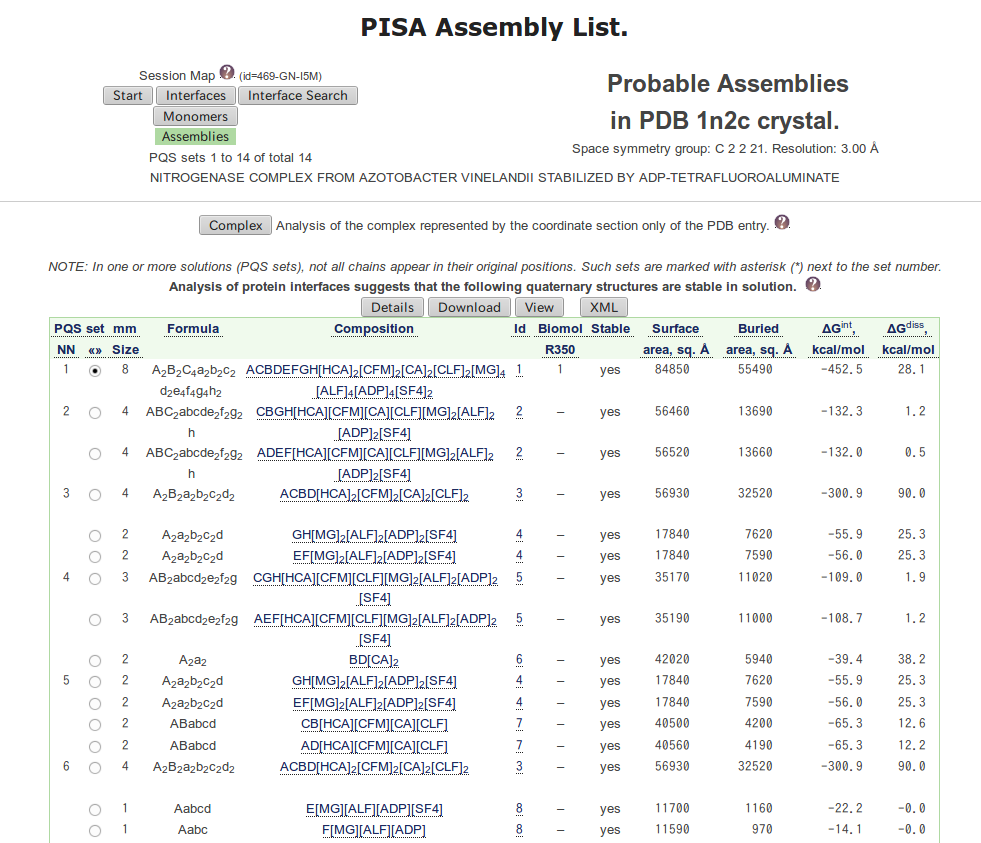
2.2.3. Assemblies¶
タンパク質の集合状態に関する以下のような情報が得られる。
リスト中の項目とその内容を下の表にまとめた。
| 項目 | 内容 |
|---|---|
mmSize |
集合体中のモノマーの数 |
Formula |
集合体の化学組成。アルファベットはモノマーのタイプを表しており、大文字がタンパク質、小文字がリガンドである。 |
Composition |
タンパク質鎖名やリガンド名とそれらの数 |
Id |
集合体のタイプ。 |
Stable |
安定した集合体であればyes、溶液中で解離する可能性があればno |
Surface area |
溶媒と接している表面積 |
Buried area |
モノマー状態では溶媒と接しているが集合体を形成すると接しなくなる部分の表面積 |
| \(\Delta G^{int}\) | 集合体形成時に得られる溶媒和自由エネルギー |
| \(\Delta G^{diss}\) | 集合体が解離するときに必要な自由エネルギー |
図 2.10 で Id の番号をクリックすると選択した集合体における界面の情報などが詳しく分かる。
ここでは8量体である Id が 1 をクリックする。
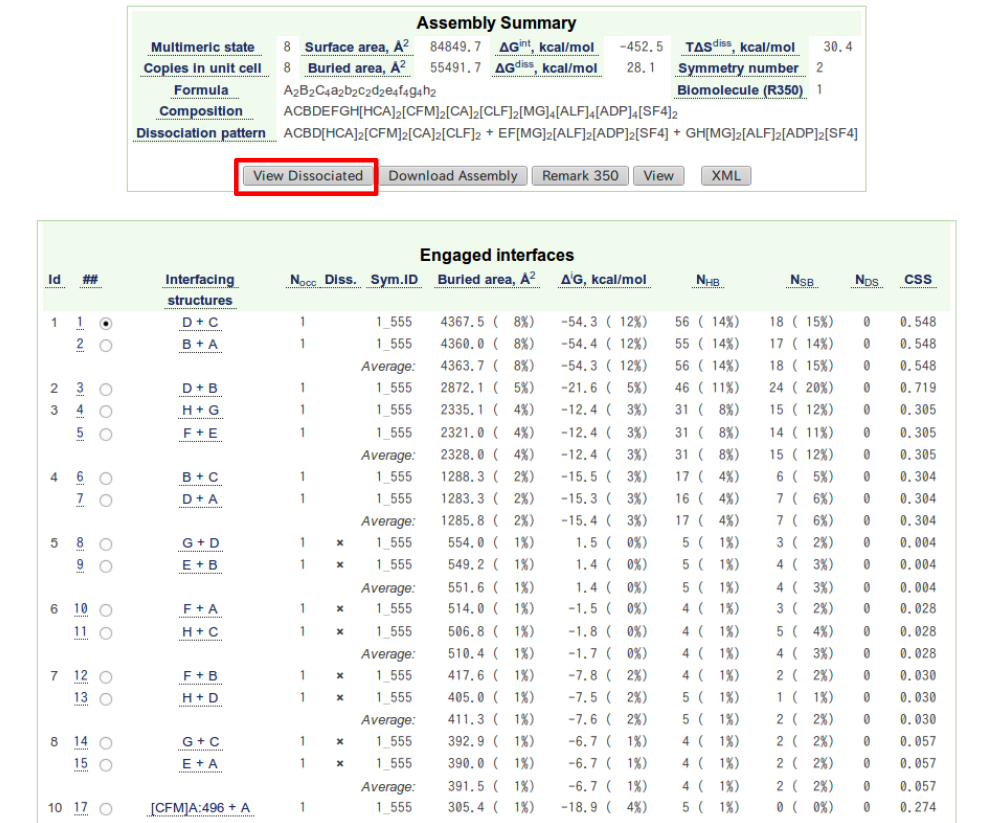
図 2.10 で View Dissociate をクリックすると、 Probable Assembly [1] が解離する際、
どのように乖離するかを図示することができる。
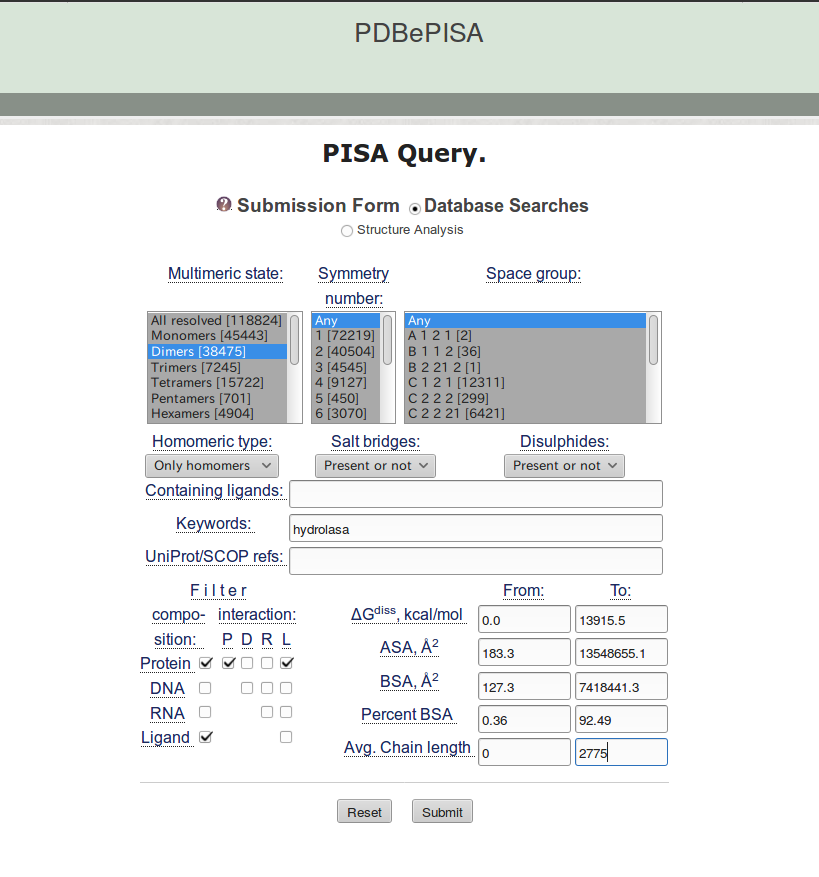
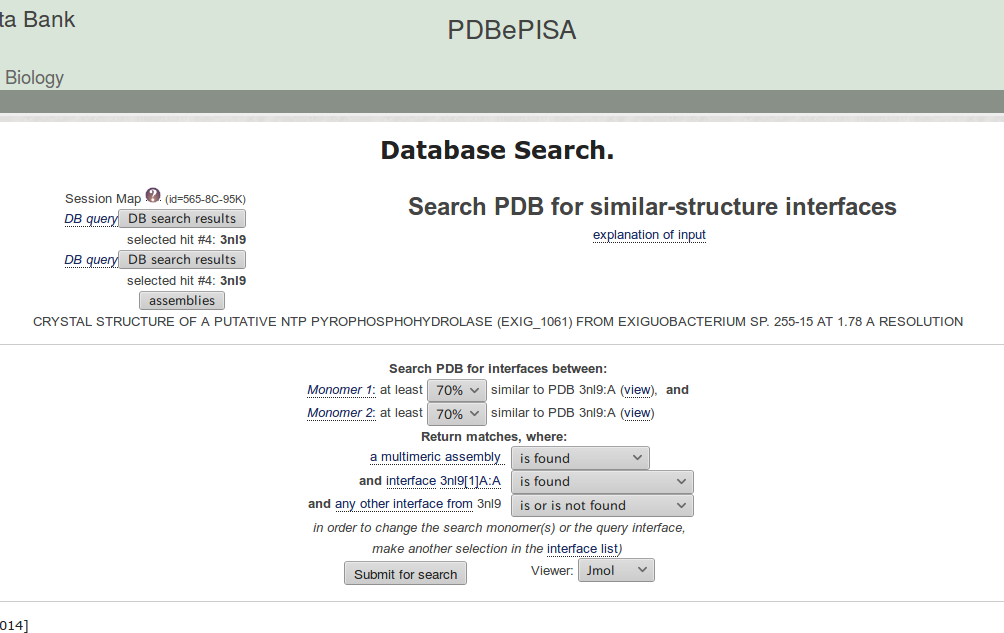
2.3. Searches による検索¶
2.3.1. 基準を満たすエントリーの検索¶
PISA はPDBの全てのエントリーのデータベースを保持しており、以下のような特定の基準に一致するエントリーを検索することができる。
- 会合数
- 対称性
- 空間群
- 塩橋やジスルフィド結合の数
- 高分子の種類
- 溶媒和自由エネルギー
例として、
同じモノマーからなる二量体でタンパク質とリガンドでできており、hydorolaseというキーワードに関連したエントリー
という条件で検索する。
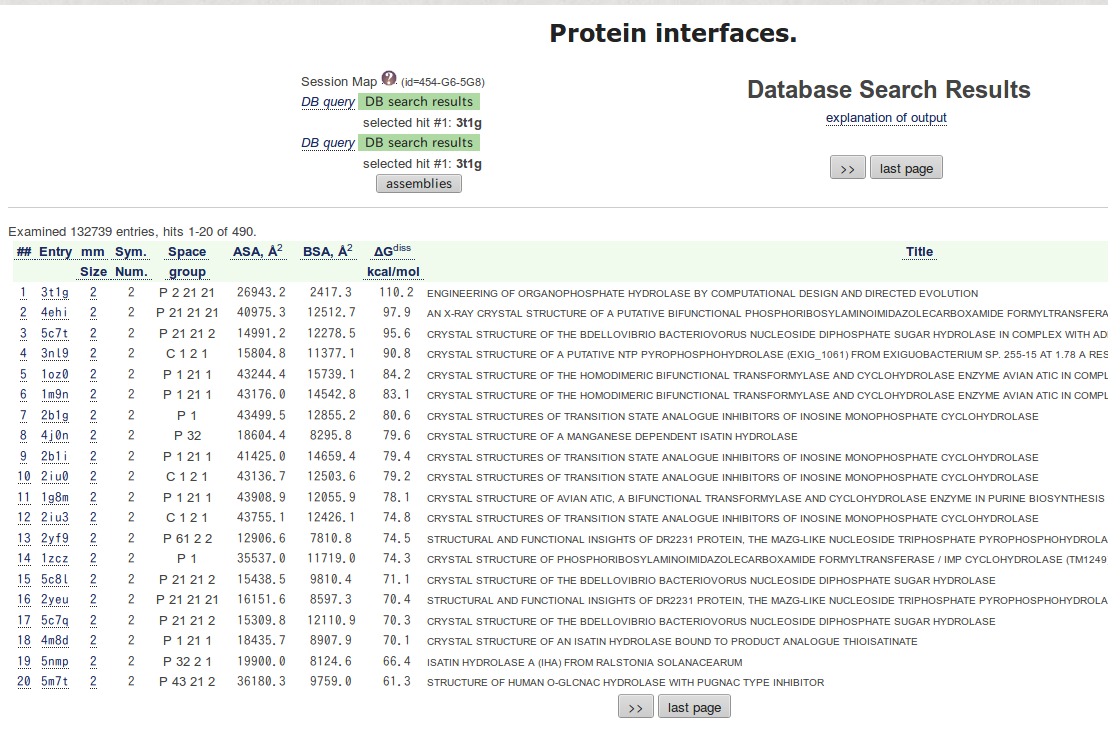
ここではエントリー 3nl9 に注目する。
図 2.14 で mmSize の列の数字 2 をクリックすると構造を表示することができる。
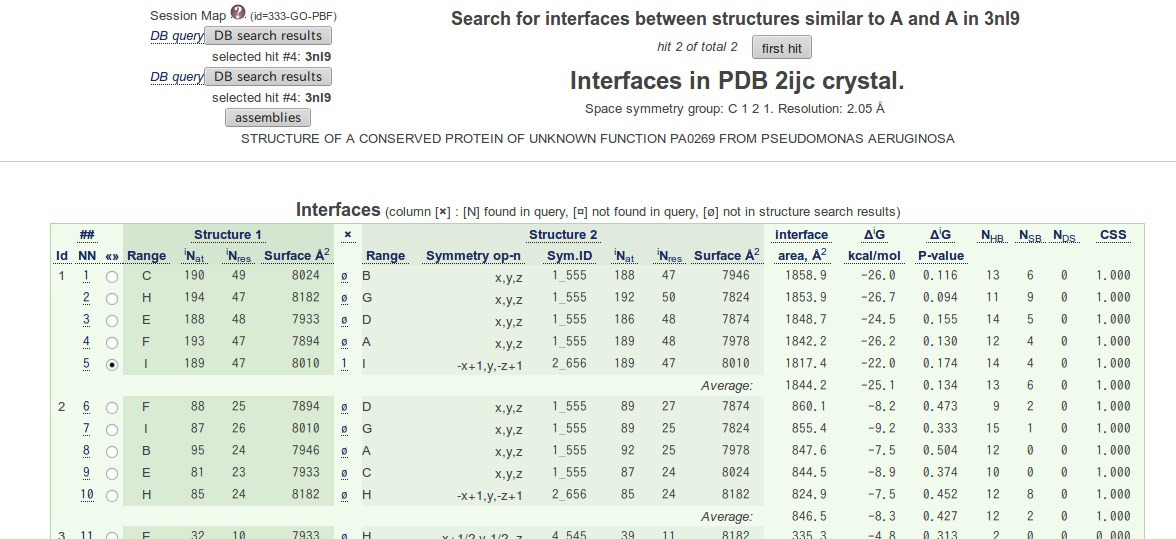
図 2.14 で Entry の列のPDB番号をクリックすると界面の詳細を見ることができる。
重要
このように、PDBの非結晶構造単位だけでなく、点群によって生成される分子も含めた 全ての可能な集合体が対象となっていることがわかる。